北极斯瓦尔巴群岛冰川流域可培养细菌的系统发育多样性及固氮菌初步研究
【类型】期刊
【作者】刘杰,李胜男,郭昱东,张丹丹,王能飞(青岛科技大学海洋科学与生物工程学院;国家海洋局第一海洋研究所)
【作者单位】青岛科技大学海洋科学与生物工程学院;国家海洋局第一海洋研究所
【刊名】极地研究
【关键词】 北极;斯瓦尔巴冰川流域;可培养细菌;潜在固氮菌;系统发育多样性;分布状况
【资助项】南北极环境综合考察与评估专项(CHINARE-2015-02-01);中央级公益性科研院所基本科研业务专项(2015T04);山东省重点研发计划(2015GSF115004)资助
【ISSN号】1007-7073
【页码】P496-505
【年份】2019
【期号】第4期
【期刊卷】7;|8;|4;|2
【摘要】从北极斯瓦尔巴群岛冰川流域的上、中、下游采集了5种代表性土壤和冰川融水样品(MT、AT、TN、BT、MS),采用常规LB和严格无氮培养基低温培养方法,对其中的可培养细菌和潜在固氮细菌进行了总菌计数以及菌株的富集、分离与纯化。经菌落菌体形态观察、16S r RNA基因初步测序排重、固氮基因nif H的PCR检测,分别选取48株可培养分离细菌和31株潜在固氮菌进行了基于16S r RNA基因全序列测定的系统发育分析。目的是初步了解该特殊生境中可培养细菌(包括潜在固氮菌)的多样性及其分布状况,为进一步研究它们在此生境中的生态功能奠定基础。实验结果表明,可培养细菌在5个采集样品中的总菌数为BT>TN>AT>MT=MS,严格无氮培养基分离菌总菌数为BT>AT>MT>TN>MS。48株可培养代表菌分属于Proteobacteria、Actinobacteria、Bacteroidetes、Firmicutes 4个门的7纲、14属之中,它们在5个样品中的多样性程度为BT(6纲、6属)>MT(4纲、6属)>AT、MS(均为3纲、3属)>TN(2纲、3属),优势菌群主要分布在γ-proteobacteria纲的Pseudomonas属(20.8%,集中在AT、BT、MS样品中)和β-proteobacteria纲的Janthinobacterium属(35.4%,集中在MS样品中),表现出一定程度的系统发育多样性。潜在固氮菌实验结果表明,能在严格无氮培养基上生长且可检测到nif H的31株代表菌具有极为单一的系统发育多样性。其中90.32%菌株集中在γ-proteobacteria纲的Pseudomonas属,仅在MT和MS样品中有3株菌分布在α-proteobacteria纲的Rhizobium属、β-proteobacteria纲的Janthinobacterium属和Actinobacteria纲的Arthrobacter属,它们与分离自其他极端环境的相关标准菌株具有高度16S r RNA基因同源性。另外,本实验还发现有8株菌的16S r RNA基因序列与相关标准菌株的相似度低于98.50%,推测它们具有成为该特殊生境下新种的潜质。
【全文】 文献传递
北极斯瓦尔巴群岛冰川流域可培养细菌的系统发育多样性及固氮菌初步研究
提要 从北极斯瓦尔巴群岛冰川流域的上、中、下游采集了5种代表性土壤和冰川融水样品(MT、AT、TN、BT、MS), 采用常规LB和严格无氮培养基低温培养方法, 对其中的可培养细菌和潜在固氮细菌进行了总菌计数以及菌株的富集、分离与纯化。经菌落菌体形态观察、16S rRNA基因初步测序排重、固氮基因 nifH的PCR检测, 分别选取48株可培养分离细菌和31株潜在固氮菌进行了基于16S rRNA基因全序列测定的系统发育分析。目的是初步了解该特殊生境中可培养细菌(包括潜在固氮菌)的多样性及其分布状况, 为进一步研究它们在此生境中的生态功能奠定基础。实验结果表明, 可培养细菌在 5个采集样品中的总菌数为BT〉TN〉 AT〉MT=MS, 严格无氮培养基分离菌总菌数为 BT 〉 AT 〉 MT 〉 TN 〉 MS。48 株可培养代表菌分属于Proteobacteria、Actinobacteria、Bacteroidetes、Firmicutes 4个门的7纲、14属之中, 它们在5个样品中的多样性程度为BT(6纲、6属)〉 MT(4纲、6属)〉 AT、MS(均为3纲、3属)〉 TN(2纲、3属), 优势菌群主要分布在 γ-proteobacteria纲的 Pseudomonas属(20.8%, 集中在 AT、BT、MS样品中)和 β-proteobacteria纲的Janthinobacterium属(35.4%, 集中在MS样品中), 表现出一定程度的系统发育多样性。潜在固氮菌实验结果表明, 能在严格无氮培养基上生长且可检测到nifH的31株代表菌具有极为单一的系统发育多样性。其中 90.32%菌株集中在 γ-proteobacteria纲的 Pseudomonas属, 仅在 MT和 MS样品中有 3株菌分布在α-proteobacteria纲的 Rhizobium属、β-proteobacteria纲的 Janthinobacterium属和 Actinobacteria纲的Arthrobacter属, 它们与分离自其他极端环境的相关标准菌株具有高度16S rRNA基因同源性。另外, 本实验还发现有8株菌的16S rRNA基因序列与相关标准菌株的相似度低于98.50%, 推测它们具有成为该特殊生境下新种的潜质。
关键词 北极 斯瓦尔巴冰川流域 可培养细菌 潜在固氮菌 系统发育多样性 分布状况
作为地球上冰川地貌的缩影, 斯瓦尔巴群岛(74°—81°N, 10°—35°E)是北极地区最大的冰川聚集区, 它不仅是全球冰川物质平衡研究的主要区域[1], 也是一个特殊的具有巨大潜力的微生物资源库[2-3]。尽管人们很早以来就对北极地区微生物进行了多种研究[4-6], 但单独针对北极冰川流域微生物群落(尤其是固氮菌等氮循环微生物)的研究还比较少见。
本研究于 2013年夏季从北极斯瓦尔巴群岛冰川流域的上、中、下游采集了5份代表性样品,采用常规 LB培养基低温培养方法对其普通类可培养细菌进行了总菌计数与分离及16S rRNA基因的系统发育多样性研究, 目的在于从海拔立体层面了解该冰川流域可培养细菌的多样性与分布状况, 为进一步研究它们在此特殊生境中的生态功能奠定基础; 同时采用严格无氮培养基低温培养方法, 对样品中的潜在固氮菌进行了总菌计数、富集分离、固氮基因 nifH-PCR检测、以及16S rRNA基因的系统发育多样性初步分析, 目的在于为后续开展的冰川区域氮循环微生物的研究提供前期经验。
1 材料与方法
1.1 样品采集
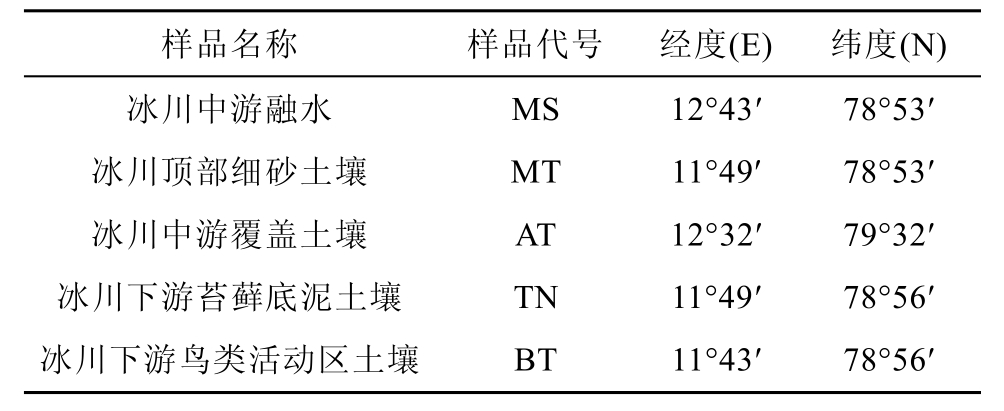
5份样品于2013年7月采集自北极斯瓦尔巴群岛冰川流域(78°53′—79°32′N, 11°43′—12°43′E),样品信息见表 1。样品采集后置于无菌离心管中4℃暂时保存, 带回实验室后立即进行菌株的分离, 剩余样品置于-80℃冰箱低温长期保藏。
表1 样品采集信息
Table 1. Information of collected samples
?
1.2 菌株分离与培养
1.2.1 培养基
1. LB培养基: 酵母提取物5.0 g, 蛋白胨10.0 g,NaCl 10.0 g, 去离子水1 000 mL, pH=7.2(固体培养基附加琼脂粉 15 g·L–1)。
2. 严格无氮培养基: 甘露醇 10.0 g, KH2PO4 0.2 g, MgSO4·7H2O 0.2 g, NaCl 0.2 g, K2SO4·2H2O或 CaSO4·2H2O 0.1 g, CaCO3 5.0 g, 去离子双蒸水1 000 mL, pH=7.2—7.4。固体培养基附加高纯度琼脂糖(西班牙 BIOWEST 产品, Agarose G-10)18.0 g·L–1。所用器皿均用 0.1 mol·L–1 HCl浸泡过夜, 再用去离子双蒸水清洗晾干后备用。
1.2.2 样品处理、总菌计数及菌株分离
考虑斯瓦尔巴群岛夏季平均气温(15℃左右)和冰川海拔, 本实验对分离菌的培养温度均设定为低温12℃。
1. 可培养细菌的计数与分离: 称取土壤样品5 g(或冰川融水5 mL), 加到含45 mL无菌生理盐水(0.85%, NaCl)和灭菌玻璃珠的三角瓶中, 充分振荡后静置20 min, 取适量上清液用无菌生理盐水进行 10倍系列梯度稀释(每组设 3次重复),涂布于LB固体平板上12℃培养7—15 d进行菌落计数, 同时根据菌落形态、颜色等特征挑取单菌落进行平板划线纯化。
2. 潜在固氮菌的计数、富集与分离: 5 g土样(或5 mL冰川融水)加到45 mL严格无氮液体培养基中于 12℃摇床 130 r·min–1富集培养, 第 7 天时转接一次新鲜培养基, 继续12℃摇床培养7—15 d。取适量上清液(根据情况适量稀释)涂布于严格无氮固体培养基平板上 12℃培养 7—15 d, 随机挑取不同形态单菌落进行平板划线纯化。总菌计数与可培养细菌方法相同, 只是培养基更换为严格无氮培养基。考虑到土壤样品中可能存在不同形式的氮源干扰, 故本次计数、分离的固氮菌株被称为潜在固氮菌。
上述两类纯化菌株经革兰氏染色镜检合格后,制成30%甘油管保藏于–80℃超低温冰箱。
1.3 细菌总DNA的提取
采用细菌总DNA碱法小量提取方法[7], 并略作修改。
1.4 固氮基因(nifH)的PCR检测
采用两对通用引物对分离的潜在固氮菌进行nifH的PCR相互印证扩增检测。
1. nifH引物对1: 是对GenBank中部分已有nifH基因进行比较分析后, 选取比较保守区域设计 的 兼 并 引 物 (nifH-100F: 5′-GGC TGC GAT CC(AGC) AAG GCC GA(CT) TC(AGC) ACC CG-3′, nifH-100R: 5′-CTG (AGC)GC CTT GTT(CT)TC GCG GAT (GC)GG CAT GGC-3′)。PCR 反应体系(50μL): 10x Taq Buffer 5μL, MgCl2(25 mM)4μL, dNTP (2 mM) 1μL, 引物对(10μM)各 1μL,基因组 DNA 1μL, Taq 酶(500 U) 0.8μL, 加 ddH2O至 50 μL。PCR 反应条件: 95℃预变性, 5 min; 94℃变性1 min; 55℃复性1 min; 72℃延伸1 min, 共30个循环; 最后72℃延伸10 min。特征扩增条带大小320 bp。
2. nifH引物对2: 参考Rosch等[8]的广泛兼并引物(nifH-34F: 5′-AAA GGY GGW ATC GGY AAR TCC ACC AC-3′, nifH-491R: 5′-TTG TTS GCS GCR TAC ATS GCC ATC AT-3′), 采用 Touchdown程序进行PCR。PCR反应体系同nifH引物对1的扩增。PCR反应条件: 95℃预变性, 5 min,95℃变性40 s, 55℃复性30 s, 72℃延伸1 min, 2个循环; 95℃变性40 s, 54℃复性30 s, 72℃延伸1 min,2个循环; 95℃变性40 s, 53℃复性30 s, 72℃延伸1 min, 2个循环; 95℃变性40 s, 52℃复性30 s,72℃延伸1 min, 2个循环; 95℃变性40 s, 51℃复性30 s, 72℃延伸1 min, 30个循环; 最后72℃总延伸6 min。特征扩增条带大小432 bp。
上述两对引物的PCR扩增产物均用1.8%琼脂糖凝胶电泳进行检测和拍照。
1.5 16S rDNA-PCR扩增及序列分析
分离菌株的16S rDNA的PCR采用通用引物(27F: 5′-AGAGTTTGATCCTGGCTCAG-3′, 1541R:5′-AAGGAGGTGATCCAGCCGCA-3′)。PCR 反应体系同nifH引物对1的扩增。PCR反应条件: 94℃预变性4 min, 94℃变性1 min, 55℃复性1 min,72℃延伸 1.5 min, 共 30个循环; 最后72℃延伸10 min。PCR扩增产物经1.2%琼脂糖凝胶电泳检测后, 再用胶回收试剂盒(天根生物技术公司产品)纯化, 送上海生工生物技术公司进行双向全长序列测定。所获序列首先用DNAstar软件进行差错整理和拼接, 然后在 EzTaxon网站(韩国)与相似性最高标准模式菌株进行比对, 再从GenBank(NCBI网站)中下载相关标准菌株序列,最后用 Clustal X 和 TreeconW 软件(采用Neighbor-joining方法)进行系统发育树的构建。
2 结果与分析
2.1 样品总菌计数
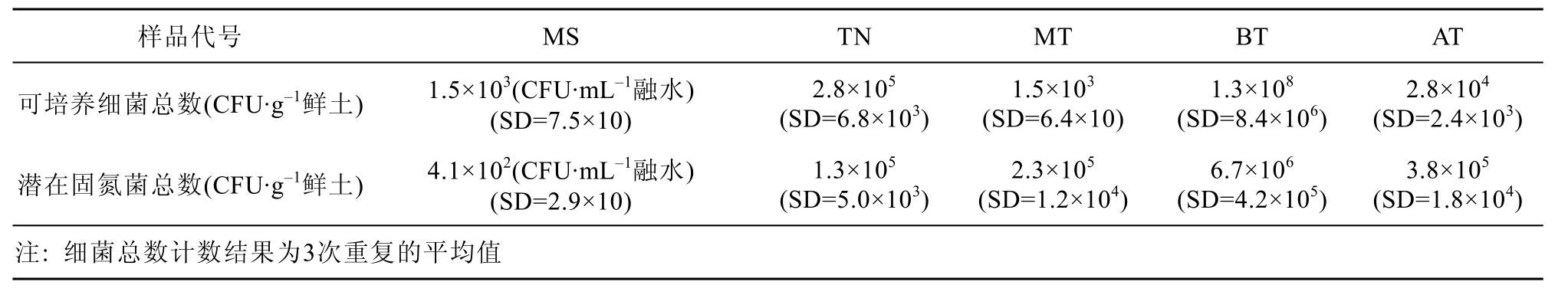
5个采集样品可培养细菌、潜在固氮菌的总菌计数结果见表2。
表2 样品中可培养细菌、潜在固氮菌总菌计数
Table 2. Population count of culturable bacteria and potential nitrogen-fixing bacteria in samples
?
表2显示, 5个样品中可培养细菌的总数含量顺序是: BT(冰川下游鸟类活动区土壤)〉TN(冰川下游苔藓底泥土壤)〉AT(冰川中游覆盖土壤)〉MT(冰川顶部细砂土壤)、MS(冰川中游融水);5个样品中潜在固氮菌总数含量顺序是: BT〉AT〉MT 〉 TN 〉 MS。
2.2 一般性可培养细菌的16S rDNA系统发育分析
经过菌落、菌体形态(包括革兰氏染色)观察、以及16S rDNA 初步单向测序排重, 从可培养分离菌中选取48株代表菌进行了16S rDNA双向全长序列测定(NCBI接受号: KU671159—KU 671206), 系统发育多样性分析结果如图1所示。
总体上看, 48株可培养细菌共分布在变形杆菌(Proteobacteria)、放线菌(Actinobacteria)、拟杆菌(Bacteroidetes)、厚壁菌(Firmicutes)等 4门、7纲、14属之中, 它们分别是Alphaproteobacteria(α-变形菌纲)的Rhizobium属(1株, 2.08%), Betaproteobacteria(β-变形菌纲)的 Polaromonas属(1株,2.08%)、Janthinobacterium 属(10株, 20.84%),Gammaproteobacteria(γ-变形菌纲)的 Pseudomonas属(17株, 35.42%), Bacilli(杆菌纲)的 Staphylococcus属(1株, 2.08%)、Macrococcus属(1株,2.08%)、Planococcus属(1株, 2.08%), Actinobacteria(放线菌纲)的 Kocuria属(2株, 4.17%)、Streptomyces属(1株, 2.08%)、Arthrobacter属(7株 14.59%)、Rhodococcus属(1株, 2.08%)、Micrococcus属(1株, 2.08%), Sphingobacteria(鞘氨醇杆菌纲)的 Pedobacter属(2株, 4.17%), 以及Flavobacteria(黄杆菌纲)的Flavobacterium属(2株,4.17%)。其中分布于 β-proteobacteria 纲、Janthin-obacterium属的10株菌(35.4%, 集中在MS样品)和γ-proteobacteria纲、Pseudomonas属的17株菌(20.8%, 集中在 AT、BT、MS 样品)为优势菌群(共占48个分析菌株的56.25%)。
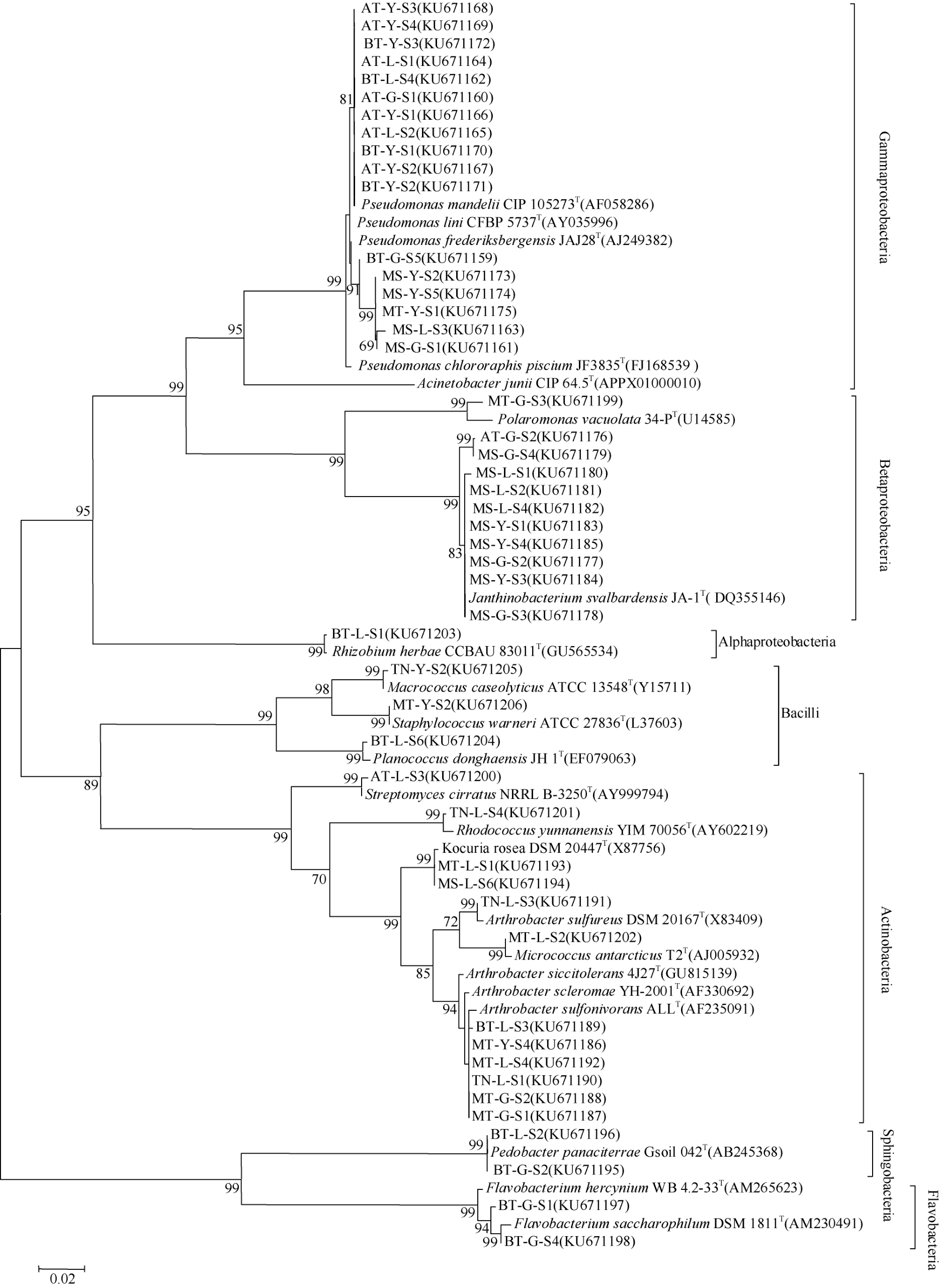
图1 可培养细菌的16S rDNA系统发育树
Fig.1. The 16S rDNA phylogenetic tree of culturable bacteria
从各菌群在样品中的分布来看, 分离自冰川顶部细砂土壤(MT)的 9株菌分布在 4纲(β-proteobacteria、Bacilli、Actinobacteria、γ-proteobacteria)、6 属(Polaromonas、Staphylococcus、Kocuria、Micrococcus、Arthrobacter、Pseudomonas), 冰川下游苔藓底泥土壤(TN)的 4株菌分布在 2纲(Bacilli、Actinobacteria)、3 属(Macrococcus、Rhodococcus、Arthrobacter), 冰川中游覆盖土壤(AT)的 9株菌分布于 3纲(Actinobacteria、β-proteobacteria、γ-proteobacteria)、3 属(Streptomyces、Janthinobacterium、Pseudomonas), 冰川中游融水(MS)的 14株菌分布于 3纲(Actinobacteria、β-proteobacteria、γ-proteobacteria)、3 属(Kocuria、Janthinobacterium、Pseudomonas), 冰川下游鸟类活动区土壤(BT)的 12株菌分布于 6纲(γ-proteobacteria、α-proteobacteria、Bacilli、Actinobacteria、Sphingobacteria、Flavobacteria)、6 属(Pseudomonas、Rhizobium、Planococcus、Arthrobacter、Pedobacter、Flavobacterium)。综合来看, 5个样品中可培养细菌的多样性顺序为: BT(6纲6属)〉MT(4纲 6属)〉 AT、MS(均为 3纲 3属)〉 TN(2纲3 属)。
从样品总菌数与多样性比较来看, BT样品的总菌数(1.3×108 CFU·g–1)和多样性(6 纲、6 属)为 5个样品中最高; TN样品总菌数中等(2.8×105 CFU·g–1), 但多样性较低(2纲、3属); AT样品总菌数(2.8×104 CFU·g–1)和多样性(3 纲、3 属)中等;而 MS和 MT样品总菌数在 5个样品中最少(1.5×103 CFU·mL–1, 1.5×103 CFU·g–1), 但仍具有一定的多样性(3纲3属, 4纲6属)。
上述结果表明, 此次采集的北极斯瓦尔巴群岛冰川流域的样品中, 普通可培养细菌具有一定程度的系统发育多样性。
2.3 潜在固氮菌的nifH-PCR检测及16S rDNA系统发育分析
经过菌落、菌体形态(包括革兰氏染色)观察初步排重, 从严格无氮培养基分离物中选取了43株菌, 采用两对引物对其进行固氮基因 nifH的PCR验证检测, 见图2。
经PCR扩增检测发现, 这43株菌中有31株能够被两对引物单独或同时扩增出比较明显的nifH特征条带(数据统计表省略)。因此, 这31株菌为代表进行了16S rDNA全长序列测定及系统发育分析。

图2 部分潜在固氮菌的nifH –PCR. a)引物对1的PCR, 特征条带大小320 bp; b)引物对2的PCR, 特征条带大小432 bp
Fig.2. nifH-PCR of part potential nitrogen-fixing bacteria. a) PCR of primer 1, the size of characteristic band is 320 bp; b)PCR of primer 2 , the size of characteristic band is 432 bp
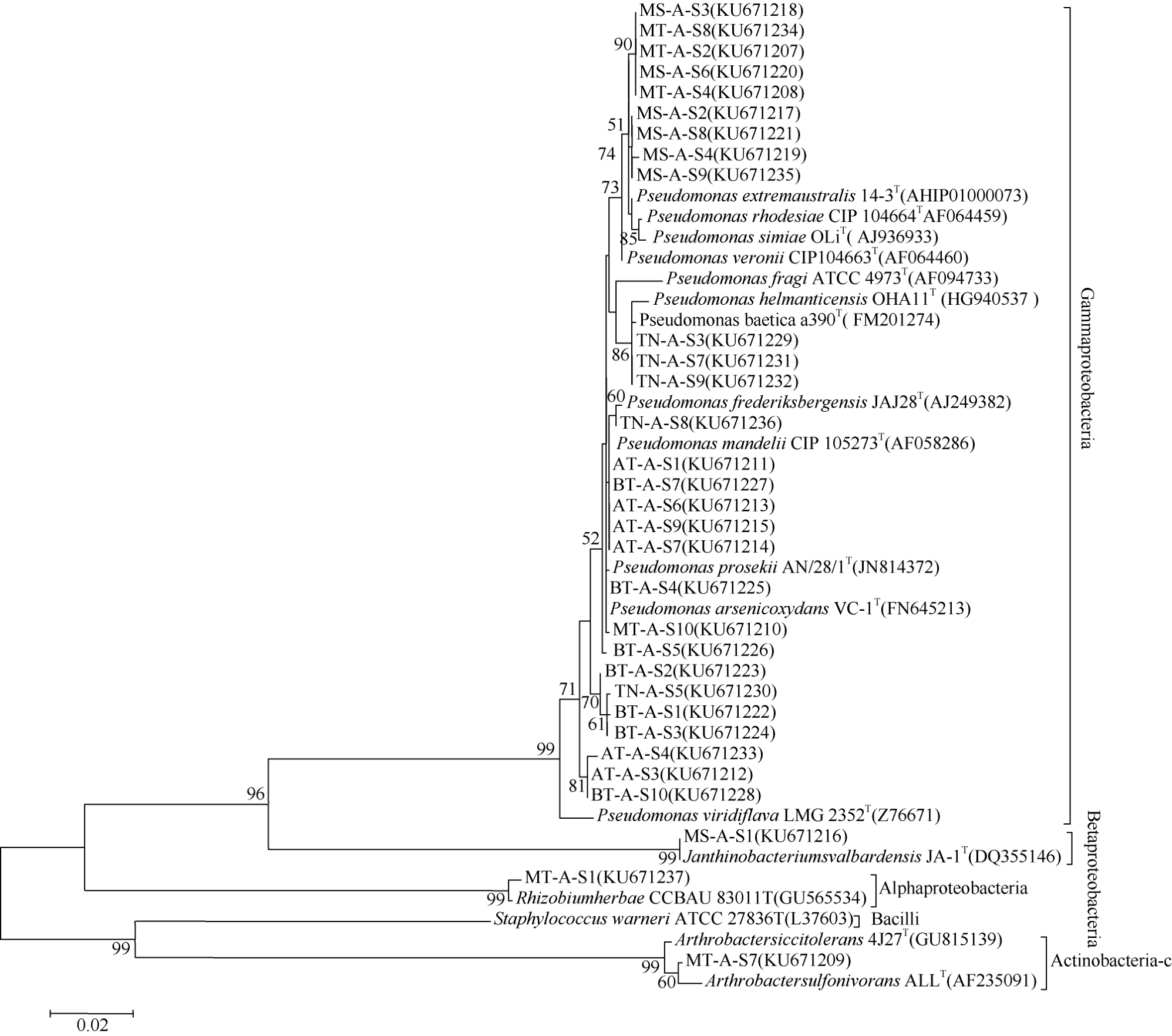
图3 含nifH潜在固氮菌的16S rDNA系统发育树
Fig.3. 16S rDNA phylogenetic tree of potential nitrogen-fixing bacteria with nifH
从图3可见, 31株潜在固氮菌中(在NCBI的16S rDNA 序列登记号为KU671207—KU671237)有 28株(占 31株代表菌株的 90.32%)集中在γ-proteobacteria 纲的 Pseudomonas属, 为绝对优势菌群, 仅MT样品中的MT-A-S7、MT-A-S1和MS样品中的MS-A-S1三株菌分别分布在Actinobacteria纲的 Arthrobacter属、α-proteobacteria纲的Rhizobium属和β-proteobacteria纲的Janthinobacterium属中。
上述结果表明, 此次从斯瓦尔巴群岛冰川流域分离的潜在固氮菌具有极为单一的系统发育多样性。
3 讨论
特殊生境必定蕴藏着与之相匹配的独特微生物多样性与种质资源。本研究从上、中、下游对北极斯瓦尔巴群岛冰川流域的可培养细菌和潜在固氮菌的系统发育多样性进行了初步分析, 并获得了如下认识。
3.1 样品含菌量分布状况
从总菌数在样品中的分布来看, 冰川下游鸟类活动区土壤(BT样品)中无论是可培养细菌还是潜在固氮菌的总菌数均为最多, 而冰川中游融水(MS)最少; 冰川顶部细砂土壤(MT)中可培养菌数量很少, 但却含有一定数量的潜在固氮菌。冰川中游覆盖土壤(AT)的可培养菌不是太多, 但潜在固氮菌数量却较多(多于MT和TN); 而冰川下游苔藓底泥(TN)的可培养菌较多, 但潜在固氮菌则很少(仅多MS)。呈现出有机质含量丰富样品中可培养菌数多而潜在固氮菌数少、有机质含量较少样品中潜在固氮菌数多但可培养菌数少的趋势。
3.2 可培养细菌多样性在样品中的分布状况
本实验不仅反映出斯瓦尔巴冰川流域的可培养细菌具有一定程度系统发育多样性(4门、7纲、14属), 而且反映出其在该冰川海拔立体层面不同生态样品中的实际分布状况。如多样性在5个采集样品中的分布程度为下游 BT样品(6纲 6属)〉上游MT样品(4纲6属)〉中游AT、MS样品(均为3纲3属)〉下游TN样品(2纲3属), 且56.25%的优势菌群主要集中在 β-proteobacteria纲和γ-proteobacteria纲。其中β-proteobacteria纲菌主要分布在 MT、AT、MS样品中, γ-proteobacteria纲菌主要分布在MT、AT、MS、BT样品中, Bacilli纲菌主要分布在 MT、TN、BT样品中, Actinobacteria纲菌主要分布在MT、TN、MS、BT样品中, 而 α-proteobacteria、Sphingobacteria、Flavobacteria纲菌则仅出现在BT样品中。值得注意的是,冰川顶部细砂土壤(MT样品)在5个样品中也有相对较高的多样性。
另外, 菌株之间的系统发育相似性在一定程度上可以反映出微生物之间的生物地理学信息。本研究中有 17株可培养细菌在 γ-proteobacteria纲内, 与其关系最近的标准模式菌株 Pseudomonas Frederiksbergensis JAJ28T(分离自丹麦哥煤气化田土壤[9])、Pseudomonas mandelii CIP 105273T(分离自天然矿泉水[10])、Pseudomonas lini CFBP 5737T(分离自法国第戎亚麻根际土壤[11])、Pseudomonas chlororaphis piscium JF3835 T(分离自瑞士淡水鱼内脏[12])的16S rDNA序列相似性在97.89%—99.72%范围。在 β-proteobacteria纲内,有8株菌与Janthinobacterium svalbardensis JA-1T(分离自斯匹次卑尔根岛冰川[13])的相似性在98.49%—99.93%范围; 有 1株菌与 Polaromonas vacuolata 34-PT(分离自南极海洋[14])的相似性为97.60%; 在 α-proteobacteria纲内, 有 1株菌与Rhizobium herbae CCBAU 83011T(分离自中国野生豆科植物根瘤[15])的相似性为99.62%。在Bacilli纲内, 有3株菌与Staphylococcus warneri ATCC 27836T(分离自人体皮肤[16])、Macrococcus caseolyticus ATCC 13548T(分离自牛奶[17])、Planococcus donghaensis JH 1T(分离自韩国东海近海沉积物[18])的相似性分别为99.86%、98.61%、98.46%。在Actinobacteria纲内, 有12株菌与Arthrobacter scleromae YH-2001T(分离自患者皮肤[19])、Rhodococcus yunnanensis YIM 70056T(分离自中国云南森林土壤[20])、Streptomyces cirratus NRRL B-3250T、Kocuria rosea DSM 20447T、Arthrobacter sulfurous DSM 20167T、Micrococcus antarcticus T2T(分离自南极长城站[21])、Arthrobacter siccitolerans 4J27T、Arthrobacter sulfonivorans ALLT的序列相似性在 98.37%—99.79%。除此之外, 在Sphingobacteria纲中, 有 2株菌与 Pedobacter panaciterrae Gsoil 042T(分离自韩国人参种植土壤[22])的序列相似性分别为 98.93%、98.59%; 在Flavobacteria纲中, 有 2株与 Flavobacterium hercynium WB 4.2-33T(分离自德国淡水河[23])及Flavobacterium saccharophilum DSM 1811T的序列相似性分别为98.07%、98.86%。
以上这种可培养细菌在斯瓦尔巴群岛冰川流域的多样性, 以及与其高度相关标准模式菌株之间(分别来自其他陆地、海洋等环境)在生态地理、生态功能、遗传进化上存在什么样的关系, 值得进一步深入研究。
3.3 对固氮菌初步研究的认识
尽管本实验采用了严格无氮培养基低温培养法和nifH-PCR检测法, 在一定程度上可以推断分离菌的固氮性质, 但仍无法最终判定它们是否具有实际固氮功效(需采用传统乙炔还原法才能确认), 因此本实验所针对的仅是一些具有潜在固氮功能的菌株, 但推测大多数具有实际固氮功效的菌株也应该存在其中。该实验对后续相关研究可以起到一定参考作用。
从多样性结果来看, 此次分离的潜在固氮菌在系统发育多样性方面表现的极为单一(90.32%集中在Pseudomonas属), 5个样品中均有分布。另外,它们与相关标准模式菌株的 16S rDNA相似度均较高(98.60%—99.86%), 而这些标准菌株多数分离自南极、北极、沙漠等极端环境, 如: Pseudomonas prosekii AN/28/1T(分离自南极James Ross岛[24])、Pseudomonas extremaustralis 14-3T (分离自南极地区[25])、Pseudomonas arsenicoxydans VC-1T(分离自智利阿塔卡马沙漠山谷沉积物[26])、Janthinobacterium svalbardensis JA-1T(分离自北极冰川[13])等。这种潜在固氮菌的单一系统发育多样性是由于培养条件限制所造成的假象, 还是极端环境中普遍存在的现象, 也是值得进一步比较探讨的。
另外, 本研究还发现 8株分离菌(MS-L-S3、MS-G-S1、MT-G-S3、AT-G-S2、BT-L-S6、BT-L-S3、MT-G-S1、BT-G-S1), 它们分属于Pseudomonas、Polaromonas、Macrococcus、Planococcus、Arthrobacter、Flavobacterium等常见属和非常见属, 与相应关系最近标准模式菌株的16S rDNA相似度在97.60%—98.49%。按照传统国际细菌分类通则, 16S rRNA基因序列相似度<97.0%和<95.0%分别是确定新种与新属的重要前提之一[27]。然而近年来的研究表明[28-29], 16S rRNA 基因序列相似度<98.65%的菌株均具有成为新种的潜在可能(前提是在其他方面具有显著特征)。据此, 上述这8株菌因其生境特殊, 应具备成为新种的潜在可能,值得采用基因组DNA-DNA杂交等多相分类手段进一步确定。
本研究仅就北极斯瓦尔巴群岛冰川流域普通类可培养细菌和潜在固氮细菌的多样性及分布状况做了初步分析, 结果可为南、北极地以及国内外高海拔冰川区域的相关研究提供一定参考。
参考文献
1 汪建君, 孙立广. 北极新奥尔松地区鸟粪土的分子有机地球化学研究[J]. 极地研究, 2007, 19(1): 29—37.
2 苗祯, 杜宗军, 李会荣, 等. 5株北极微藻藻际环境的细菌多样性[J]. 生态学报, 2015, 35(5): 1587—1600.
3 乔宗赟, 曾胤新, 董培艳, 等. 2011年夏季北极王湾细菌群落结构分析及浮游细菌丰度检测[J]. 极地研究, 2015, 27(3): 246—254.
4 Francis C A, Co E M, Tebo A B M. Enzymatic manganese(II) Oxidation by a marine α-proteobacterium[J]. Applied and Environmental Microbiology, 2001, 67(9): 4024—4029.
5 Le V T, Tuyen H, Helmke E, et al. Cloning of two pectate lyase genes from the marine Antarctic bacterium Pseudoalteromonas haloplanktis strain ANT/505 and characterization of the enzymes[J]. Extremophiles, 2001, 5(1): 35—44.
6 武兆宇. 北极海冰及胶州湾沉积物中细菌新型质粒的筛选、序列分析以及性质研究[D]. 济南: 山东大学, 2010.
7 赵斌, 何绍江. 微生物学实验[M]. 北京: 科学出版社, 2002.
8 Rösch C, Mergel A, Bothe H. Biodiversity of denitrifying and dinitrogen-fixing bacteria in an acid forest soil[J]. Applied and Environmental Microbiology, 2002, 68(8): 3818—3829.
9 Andersen S M, Johnsen K, Sørensen J, et al. Pseudomonas frederiksbergensis sp. nov., isolated from soil at a coal gasification site[J].International Journal of Systematic and Evolutionary Microbiology, 2000, 50(6): 1957—1964.
10 Verhille S, Baida N, Dabboussi F, et al. Taxonomic Study of Bacteria Isolated from Natural Mineral Waters: Proposal of Pseudomonas jessenii sp. nov. and Pseudomonas mandelii sp. nov[J]. Systematic and Applied Microbiology, 1999, 22(1): 45—58.
11 Delorme S, Lemanceau P, Christen R, et al. Pseudomonas lini sp. nov., a novel species from bulk and rhizospheric soils[J]. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(2): 513—523.
12 Burr S E, Gobeli S, Kuhnert P, et al. Pseudomonas chlororaphis subsp. Piscium subsp. nov., isolated from freshwater fish[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(12): 2753—2757.
13 Avguštin J A, Bertok D Ž, Kostanjšek R, et al. Isolation and characterization of a novel violacein-like pigment producing psychrotrophic bacterial species Janthinobacterium svalbardensis sp. nov[J]. Antonie van Leeuwenhoek, 2013, 103(4): 763—769.
14 Irgens R L, Gosink J J, Staley J T. Polaromonas vacuolata gen. nov., sp. nov., a psychrophilic, marine, gas vacuolate bacterium from Antarctica[J]. International Journal of Systematic Bacteriology, 1996, 46(3): 822—826.
15 Ren D W, Wang E T, Chen W F, et al. Rhizobium herbae sp. nov. and Rhizobium giardinii-related bacteria, minor microsymbionts of various wild legumes in China[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(8): 1912—1920.
16 Kloos W E, Schleifer K H. Isolation and characterization of staphylococci from human skin. II. descriptions of four new species:Staphylococcus warneri, Staphylococcus capitis, Staphylococcus hominis, and Staphylococcus simulans[J]. International Journal of Systematic Bacteriology, 1975, 25(1): 62—79.
17 Kloos W E, Ballard D N, George C G, et al. Delimiting the genus Staphylococcus through description of Macrococcus caseolyticus gen.nov., comb. nov. and Macrococcus equipercicus sp. nov., and Macrococcus bovicus sp. nov. and Macrococcus carouselicus sp. nov.[J].International Journal of Systematic Bacteriology, 1998, 48(3): 859—877.
18 Choi J H, Im W T, Liu Q M, et al. Planococcus donghaensis sp. nov., a starch-degrading bacterium isolated from the East Sea, South Korea[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(11): 2645—2650.
19 Huang Y, Zhao N X, He L, et al. Arthrobacter scleromae sp. nov. isolated from human clinical specimens[J]. Journal of Clinical Microbiology, 2005, 43(3): 1451—1455.
20 Zhang Y Q, Li W J, Kroppenstedt R M, et al. Rhodococcus yunnanensis sp. nov., a mesophilic actinobacterium isolated from forest soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(3): 1133—1137.
21 Liu H C, Xu Y, Ma Y H, et al. Characterization of Micrococcus antarcticus sp. nov., a psychrophilic bacterium from Antarctica[J].International Journal of Systematic and Evolutionary Microbiology, 2000, 50(2): 715—719.
22 Yoon M H, Ten L N, Im W T, et al. Pedobacter panaciterrae sp. nov., isolated from soil in South Korea[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(2): 381—386.
23 Cousin S, Pauker O, Stackebrandt E. Flavobacterium aquidurense sp. nov. and Flavobacterium hercynium sp. nov., from a hard-water creek[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57: 243—249.
24 Kosina M, Barták M, Mašlaňová I, et al. Pseudomonas prosekii sp. nov., a novel psychrotrophic bacterium from Antarctica[J]. Current Microbiology, 2013, 67(6): 637—646.
25 Tribelli P M, Iustman L J R, Catone M V, et al. Genome sequence of the polyhydroxybutyrate producer Pseudomonas extremaustralis, a highly stress-resistant antarctic bacterium[J]. Journal of Bacteriology, 2012, 194(9): 2381—2382.
26 Campos V L, Valenzuela C, Yarza P, et al. Pseudomonas arsenicoxydans sp nov., an arsenite-oxidizing strain isolated from the Atacama desert[J]. Systematic and Applied Microbiology, 2010, 33(4): 193—197.
27 Stackebrandt E, Boebel B M. Taxonomic Note: A place for DNA-DNA reassociation and 16S rRNA Sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic Bacteriology, 1994, 44(4): 846—849.
28 Cho J C, Tiedje J M. Bacterial species determination from DNA-DNA hybridization by using genome fragments and DNA microarrays[J]. Applied and Environmental Microbiology, 2001, 67(8): 3677—3682.
29 Kim M, Oh H S, Park S C, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2):346—351.
PRELIMINARY STUDY ON PHYLOGENETIC DIVERSITY OF CULTURABLE BACTERIA AND NITROGEN-FIXING BACTERIA IN THE SVALBARD GLACIERS OF THE ARCTIC
Abstract Five different samples of representative soil and meltwater (MT, AT, TN, BT, MS) were collected from the upstream, midstream and downstream of the Arctic Svalbard glaciers. The culturable bacteria and potential nitrogen-fixing bacteria were counted, enriched and isolated by using LB medium and strict nitro-gen-free medium at the low temperature, respectively. After colony observation, preliminary 16S rRNA gene sequencing and nifH-PCR, 48 culturable bacterial strains and 31 potential nitrogen-fixing bacterial strains were selected to examine the phylogenetic diversity based on the almost full-length 16S rRNA gene sequences. The purpose was to obtain a preliminary understanding of the diversity and distribution of these bacteria in this special area, and to provide a basis for further research on their ecological functions. The results showed that the populations of the culturable bacteria in the five samples were ordered as BT 〉 TN 〉AT 〉 MT=MS. The populations of potential nitrogen-fixing bacteria were ordered as BT 〉 AT 〉 MT 〉 TN 〉MS. Forty-eight culturable bacteria belonged to 4 phyla (Proteobacteria, Actinobacteria, Bacteroidetes, Firmicutes), 7 classes and 14 genera, and their degrees of diversity were ordered as BT(6 classes, 6 genera)〉MT(4 classes, 6 genera)〉 AT=MS(3 classes, 3 genera)〉 TN(2 classes, 3 genera). Meanwhile, the majority of culturable bacterial isolates were composed of Pseudomonas of γ-proteobacteria (20.8%, isolated from the AT, BT and MS samples) and Janthinobacterium of β-proteobacteria(35.4%, isolated from the MS sample).The experimental results of the potential nitrogen-fixing bacteria showed that the diversity of 31 strains isolated from the five samples was very low. Of the strains, 90.32% belonged mainly to Pseudomonas(γ-proteobacteria), only 3 strains isolated from the MT and MS samples belonged to Rhizobium(α-proteobacteria), Janthinobacterium (β-proteobacteria) and Arthrobacter (Actinobacteria C), respectively.There was high 16S rRNA gene homology among these potential nitrogen-fixing strains and some related standard-type strains isolated from other extreme environment. In addition, eight strains that showed less than 98.6% 16S rRNA gene-sequence similarity with strains of related type may represent potential novel species in this special ecological environment.
Key words Arctic, Svalbard glaciers, culturable bacteria, potential nitrogen-fixing bacteria, phylogenetic diversity, distribution
doi: 10. 13679/j. jdyj. 2017. 4. 496
[收稿日期] 2016年5月收到来稿, 2016年11月收到修改稿
[基金项目] 南北极环境综合考察与评估专项(CHINARE-2015-02-01)、中央级公益性科研院所基本科研业务专项(2015T04)和山东省重点研发计划(2015GSF115004)资助
