崇测冰帽不同的冰川样品可培养细菌群落结构差异
【类型】期刊
【作者】张淑红,侯书贵,包格日乐,许浩,刘亚平,李治国(商丘师范学院生命科学学院;南京大学地理与海洋学院;中国科学院寒区旱区环境与工程研究所冰冻圈科学国家重点实验室;商丘师范学院环境与规划学院)
【作者单位】商丘师范学院生命科学学院;南京大学地理与海洋学院;中国科学院寒区旱区环境与工程研究所冰冻圈科学国家重点实验室;商丘师范学院环境与规划学院
【刊名】微生物学报
【关键词】 可培养细菌;群落结构;崇测冰帽;冰川
【资助项】国家自然科学基金(31100369,41101072,41330526)
【ISSN号】0001-6209
【页码】P708-718
【年份】2019
【期号】第4期
【期刊卷】1;|7;|8;|4;|5;|2
【摘要】【目的】研究细菌群落组成在西昆仑崇测冰帽冰川雪样、冰碛物和土样中的差异。【方法】通过传统的纯培养和菌株16S r RNA基因序列鉴定,分析菌株在门水平和属水平的群落结构。【结果】冰川细菌由Actinobacteria、Firmicutes、Proteobacteria和Bacteroidetes 4个门组成。雪样以Proteobacteria为优势,而土样和冰碛物则以Actinobacteria为优势。在属的水平上,冰川土样中的优势属仅有Arthrobacter,雪样中的优势属主要有Methylobacterium、Modestobacter、Hymenobacter、Brevundimonas、Bacillus这5种。雪环境的细菌群落结构与冰碛物和土样的差异性较大,而冰碛物和土样之间的差异性不大。Skermanella可能为崇测冰帽所特有的细菌。【结论】初步说明了在冰川退缩的气候环境下,冰川雪样细菌多样性的脆弱性,以及冰川雪环境细菌资源保护的重要性。
【全文】 文献传递
微生物学报
Acta Microbiologica Sinica
2016, 56(4): 708-718
http://journals.im.ac.cn/actamicrocn DOI: 10.13343/j.cnki.wsxb.20150364
崇测冰帽不同的冰川样品可培养细菌群落结构差异
摘要:【目的】研究细菌群落组成在西昆仑崇测冰帽冰川雪样、冰碛物和土样中的差异。【方法】通过传统的纯培养和菌株16S rRNA基因序列鉴定,分析菌株在门水平和属水平的群落结构。【结果】冰川细菌由Actinobacteria、Firm icutes、Proteobacteria和Bacteroidetes 4个门组成。雪样以Proteobacteria为优势,而土样和冰碛物则以Actinobacteria为优势。在属的水平上,冰川土样中的优势属仅有Arthrobacter,雪样中的优势属主要有Methylobacterium、Modestobacter、Hymenobacter、Brevundimonas、Bacillus这5种。雪环境的细菌群落结构与冰碛物和土样的差异性较大,而冰碛物和土样之间的差异性不大。Skermanella可能为崇测冰帽所特有的细菌。 【结论】初步说明了在冰川退缩的气候环境下,冰川雪样细菌多样性的脆弱性, 以及冰川雪环境细菌资源保护的重要性。
关键词:可培养细菌,群落结构,崇测冰帽,冰川
冰川作为最大的淡水资源,覆盖了大约地球表面积的10%。高原上覆盖的冰川和雪不仅影响全球气候,也作为环境变化的敏感指示。目前对冰川微生物的研究从新生事物演变成为备受关注的科学领域,其主要内容涉及冰川生物的多样性、生态和进化及其与环境的关系,并取得了大量的研究成果。但对西昆仑地区冰川微生物的研究较少。
目前关于冰川细菌的研究通常以单一生境条件为研究对象,如通常集中在冰芯[1-2]、雪样[3]、土样[4]和冰川融水[5]等,而关于多个生境之间细菌多样性或群落的差异研究较少,仅见到Liu等的研究。首先,他们比较了珠穆朗玛峰北坡冰川融水和冰碛湖的细菌多样性和丰度,结果是最高的细菌丰度和细菌多样性都发生在海拔5152 m处开放的冰碛湖;而最低的细菌丰度和细菌多样性则发生在海拔6350 m处的冰川融水[6]。其次他们对喜马拉雅山南坡Yala冰川雪、冰碛湖、冰川溪流3种生境的细菌丰度和多样性进行了研究。冰碛湖的细菌丰度最高,而海拔5504 m处的雪样细菌丰度最低。雪样中的细菌群落与冰碛湖和冰川溪流中的细菌群落差异性很大[7]。
本文通过纯培养方法,探讨西昆仑崇测冰帽冰川雪、冰碛石、土壤三种生境中细菌群落结构的变化,为冰川微生物多样性及与其生态环境的关系提供一些知识积累。
1 材料和方法
西昆仑山有冰川5485条,冰川面积8817.78 km2,占整个昆仑山冰川总面积的3/4以上。崇测冰帽位于西昆仑山脉南侧,顶部海拔6580 m。冰川面积163.06 km2,是我国最大的冰帽。该冰川全长约7000 m,末端高度约5800 m,平衡线高度5930 m,两处突出的穹顶海拔分别为6532 m和6374 m[8]。
1.1 样品的采集
2013年10月从崇测冰帽沿着不同海拔从高到低分别采集积累区雪样、平衡线雪样、消融区雪样以及冰川末端雪样各3个。在冰川末端位点采集冰碛物样品3个。沿着冰川末端、冰川末端100、200、300、400 m各采集3个土样。每个样品采用5点梅花取样法,雪样装入采样瓶,土样去除石块等杂物,装入克林莱密实袋。所有样品置于冰柜中,以低温(≤-5 °C)方式运回实验室并置冷库中(≤-15 °C)贮存。
1.2 细菌的分离培养
1.2.1 土样和雪样的处理:在超净工作台上称取10 g土样置于盛有90 m L PBS(pH 7)溶液和玻璃珠的三角瓶内,气浴振荡器(CHA-SA)上16 °C振荡30 m in后,梯度稀释至10-3和10-4。每个梯度取200 μL涂平板。
雪样置于4 °C缓慢融化,将约400-700 m L融水用小型切向流超滤系统(Labscale,M illipore),加上0.22 μm无菌M illipore微孔滤膜,进行过滤。将滤膜悬浮于3.5 m L的PBS(pH 7)缓冲液中。振荡后取350 μL涂平板。
1.2.2 菌株分离:所用培养基为R2A、0.25R2A、0.2LB、0.5TSA。每种培养基配方具体如下。(1)R2A培养基配方(g/L):酵母提取物0.5,示蛋白胨0.5,酪蛋白氨基酸0.5,可溶性淀粉0.5,葡萄糖0.5,丙酮酸钠0.3,KH2PO40.3,M gSO4·7H2O 0.05,pH 7.2。(2) LB培养基配方(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,pH 7.0-7.2。(3)TSA培养基配方(g/L):胰蛋白胨15,大豆胨5,NaCl 30,pH 7.0-7.4。
每种培养基做6个平行,其中3个置于4 °C条件下培养15 d,另外3个置于15 °C条件下培养7 d。培养后依据细菌形态特征进行分离与纯化。由于细菌的种类不同,它所形成的菌落在形状、大小、高低、位置、表面粗细、边缘形状、色调、透明度,以及菌落质地、软硬、粘稠度等方面,也各不相同。因此,菌落是菌种鉴别上的一个重要特征。本研究根据这一理论基础,挑取不同菌落形态的菌株,纯化后的细菌于4 °C斜面保存及15%甘油-20 °C保存。
1.3 培养菌株16S rRNA基因序列分析
1.3.1 16S rRNA基因序列扩增:选取菌落形态不同的菌株,进行菌落PCR。以细菌通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-CGGTAACCTTGTTACGACTT-3′)对样品进行PCR扩增。反应体系为:1×PCR缓冲液,2.5 mmol/L MgCl2,0.2 mmol/L dNTPs,正反向引物各l0 pmol/L(上海生工生物工程有限公司合成),1 U Taq DNA聚合酶(TaKaRa,R001AM),1 μL DNA(10 ng/μL)模板,ddH2O补足至25 μL。PCR扩增条件:94 °C 2 m in;94 °C 1 min,56 °C1 m in,72 °C 1.5 m in,30个循环;72 °C 10 m in。将dd H2O用作阴性对照。扩增产物用1.0%琼脂糖凝胶电泳进行检测。
PCR产物送往上海生工生物工程有限公司进行序列分析。所获得的序列在GenBank上进行登陆,注册号为:KR263058-KR263116。
1.3.2 16S rRNA序列比较及系统发育树构建:经测序所得的序列在GenBank数据库中进行BLAST比对,选取相似性最高的已知序列下载后用于系统发育树的构建。用Clustal W 1.81[9]对序列进行对位排序,MEGA 5.02 (M olecu lar Evolutionary Genetics Analysis)软件,以邻接法(Neighbor-Joining,NJ)构建系统发育树状图,通过bootstrap法评价进化树分支聚类的稳定性,重复次数为500。
表1.采集样品的经纬度及海拔
Table 1.Latitude, longitude, altitude of samples
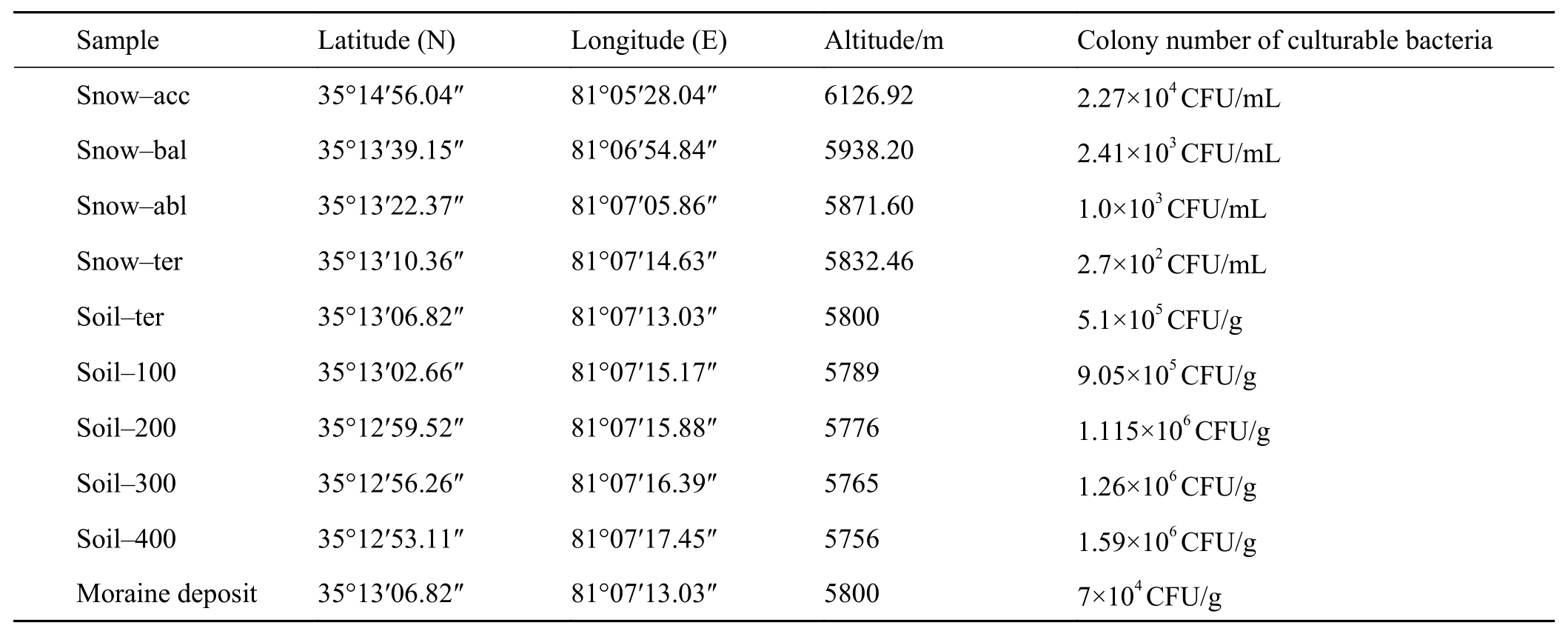
Sample Latitude (N) Longitude (E) Altitude/m Colony number of culturable bacteria Snow-acc 35°14′56.04″ 81°05′28.04″ 6126.92 2.27×104CFU/m L Snow-bal 35°13′39.15″ 81°06′54.84″ 5938.20 2.41×103CFU/m L Snow-abl 35°13′22.37″ 81°07′05.86″ 5871.60 1.0×103CFU/m L Snow-ter 35°13′10.36″ 81°07′14.63″ 5832.46 2.7×102CFU/m L Soil-ter 35°13′06.82″ 81°07′13.03″ 5800 5.1×105CFU/g Soil-100 35°13′02.66″ 81°07′15.17″ 5789 9.05×105CFU/g Soil-200 35°12′59.52″ 81°07′15.88″ 5776 1.115×106CFU/g Soil-300 35°12′56.26″ 81°07′16.39″ 5765 1.26×106CFU/g Soil-400 35°12′53.11″ 81°07′17.45″ 5756 1.59×106CFU/g Moraine deposit 35°13′06.82″ 81°07′13.03″ 5800 7×104CFU/g
2 结果和分析
崇测冰帽各冰川环境可培养细菌浓度具体如表1。该表显示,相同生境中,随着取样点海拔的降低,可培养细菌浓度基本呈现增加趋势。土样中的可培养细菌浓度最高,冰碛物次之,雪样的最低。
从土样和雪样中共恢复出92个不同菌落形态的菌株,全部用于16S rRNA序列分析和系统发育树的构建。经分析,这些序列归属于Actinobacteria、Firm icutes、Proteobacteria和Bacteroidetes 4大类。
Actinobacteria门包括Arthrobacter、Nocardioides、Kocuria、Janibacter、Microbacterium、Clavibacter、M ycetoco la、M odestobacter、Rhodococcus、Mycobacterium、Patulibacter 11个不同的属,由3个分支组成(图1)。第一大分支由21个菌株组成,包括来自冰碛物的bw2、bw1、b2w1、b2r1、by6、by2、bm 2、bw 9,来自土样的s1w 1、s4w 14、s10w 9、s7r1、s1y2、s1r2、s10w 16、s7r3、s7y2、s4r3、s7y3,以及来自雪样的x4y3和x4r5。来自雪样的菌株x4r5与来自土样的s7r3归为同一个属,即Kocuria。来自雪样的x4y3归属为Janibacter,这个属不与其它的来自土样和冰碛物的菌株相重合。第二分支由9个菌株组成,包括来自冰碛物的br1、br3、bw 4、by3和来自雪样的x4y5、x11r1、x8y1、x8a1、x4a1。在这一分支中,x8a1、bw4、by3和x4a1共同归属到了Nocardioides。其它菌株亲缘关系都比较远。第三分支仅由来自冰碛物的br2这一株菌组成。
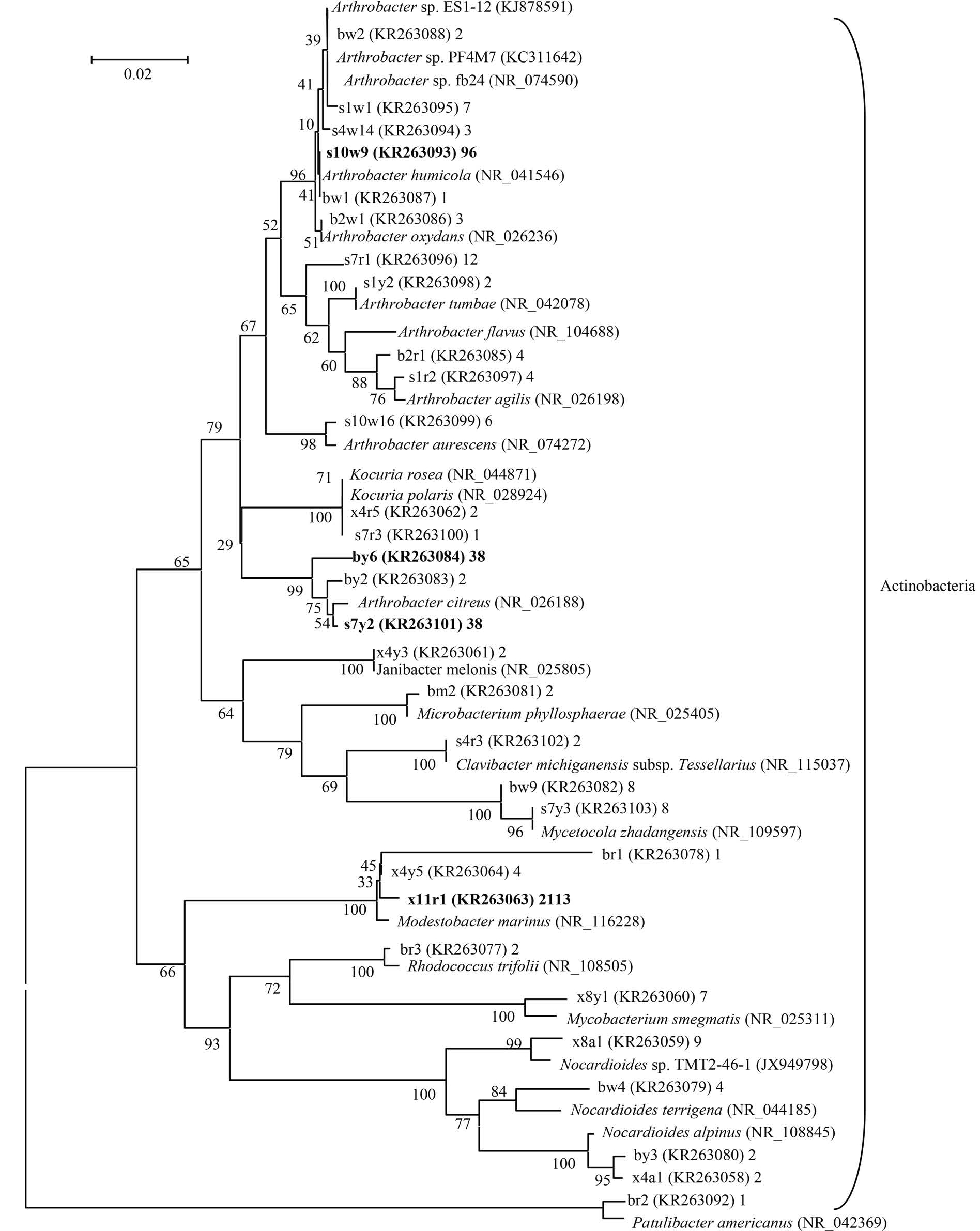
图1.西昆仑崇测冰帽可培养细菌16S rRNA基因序列Actinobacteria门的NJ系统发育树
Figure 1.Neighbor-Joining tree show ing the phylogenetic relationships of culturable bacteria from Chongce Ice Cap based on 16S rRNA gene sequences for Actinobacteria. Numbers in parentheses indicate the GenBank accession numbers. The number after each isolates’ accession number indicates the number of their colonies. Numbers at branch points are the percentage supported by bootstrap. Scale bar equals approximately 2% nucleotide divergence.
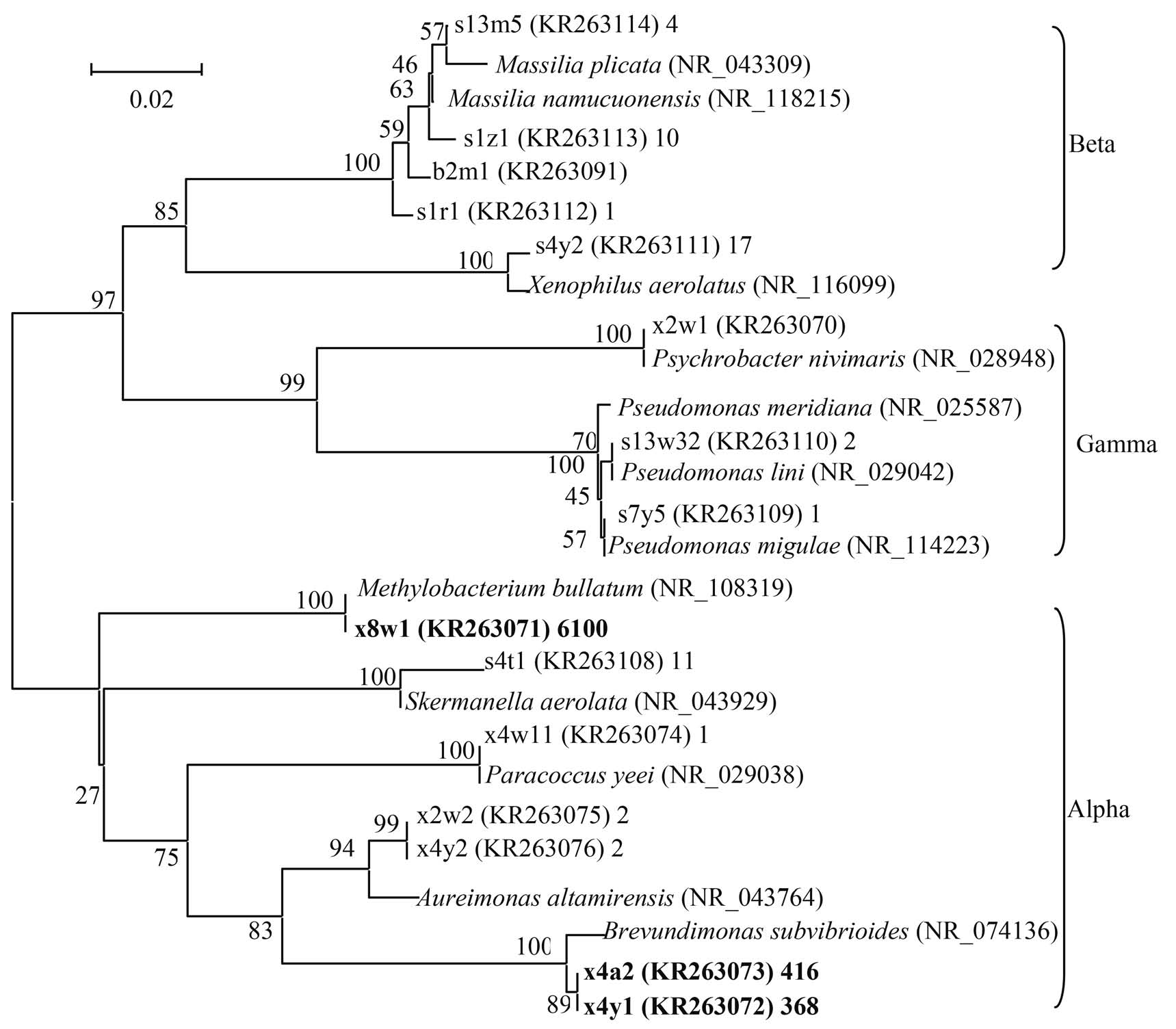
图2.西昆仑崇测冰帽可培养细菌16S rRNA基因序列Proteobacteria门的NJ系统发育树
Figure 2.Neighbor-Joining tree show ing the phylogenetic relationships of culturable bacteria from Chongce Ice Cap based on 16S rRNA gene sequences for Proteobacteria.
第二大门为Proteobacteria,由Alphaproteobacteria、Betaproteobacteria、Gammaproteobacteria 3个纲组成,包括Pseudomonas、Psychrobacter、Xenophilus、Massilia、Methylobacterium、Skermanella、Paracoccus、Aurantimonas、Brevundimonas 9个不同的属。如图2所示,来自雪样的菌株集中在Alphaproteobacteria,而来自土样的菌株集中在Betap roteobacteria和Gammaproteobacteria。在Proteobacteria类群中,没有来自冰碛物的菌株。Gammaproteobacteria由3个菌株组成,即来自土样的s7y5、s13w 32和来自雪样的x2w 1。该纲均归到Pseudom onas。Betaproteobacteria由5个菌株组成,包括来自土样的s4y2、s1r1、s1z1、s13m5,以及来自冰碛物的b 2 m 1。该纲菌株集中在M a s s i l i a属。A lphaproteobacteria由7个菌株组成,包括来自雪样的x8w1、x4w 11、x4a2、x4y1、x2w 2、x4y2,和来自土样的s4t1。在Proteobacteria门,来自雪样的菌株主要出现在A lphaproteobacteria类群,而来自土样的菌株主要出现在Betap ro teobacteria和Gammaproteobacteria类群。
第三个门是Firm icu tes,包括Bac illus、Brevibacterium、Aerococcus、Paenisporosarcina、Lysinibacillus这5个属(图3)。Firm icutes类群由3个分支组成,第一分支由5个菌株组成,包括来自雪样的x4w 12和x11w 1,以及来自土样的s10w 5、s1w8、s4w5。第二分支仅由来自冰碛物的1个菌株bw5组成。第三分支由来自雪样的x4w2和来自土样的s10m1这2个菌株组成。在Firm icutes中,Bacillus是共同存在于土样和雪样的属,其它4个属在土样、雪样和冰碛物中没有重合的。
第四个门是Bacteroidetes,由2个分支组成。第一分支由4个菌株组成,包括来自雪样的x4r6和 x4r7,来自土样的s1r3,来自冰碛物的br4。这些菌株都归属于Hymenobacter。另一分支仅由来自土样的s13y2这一株菌组成(图3)。
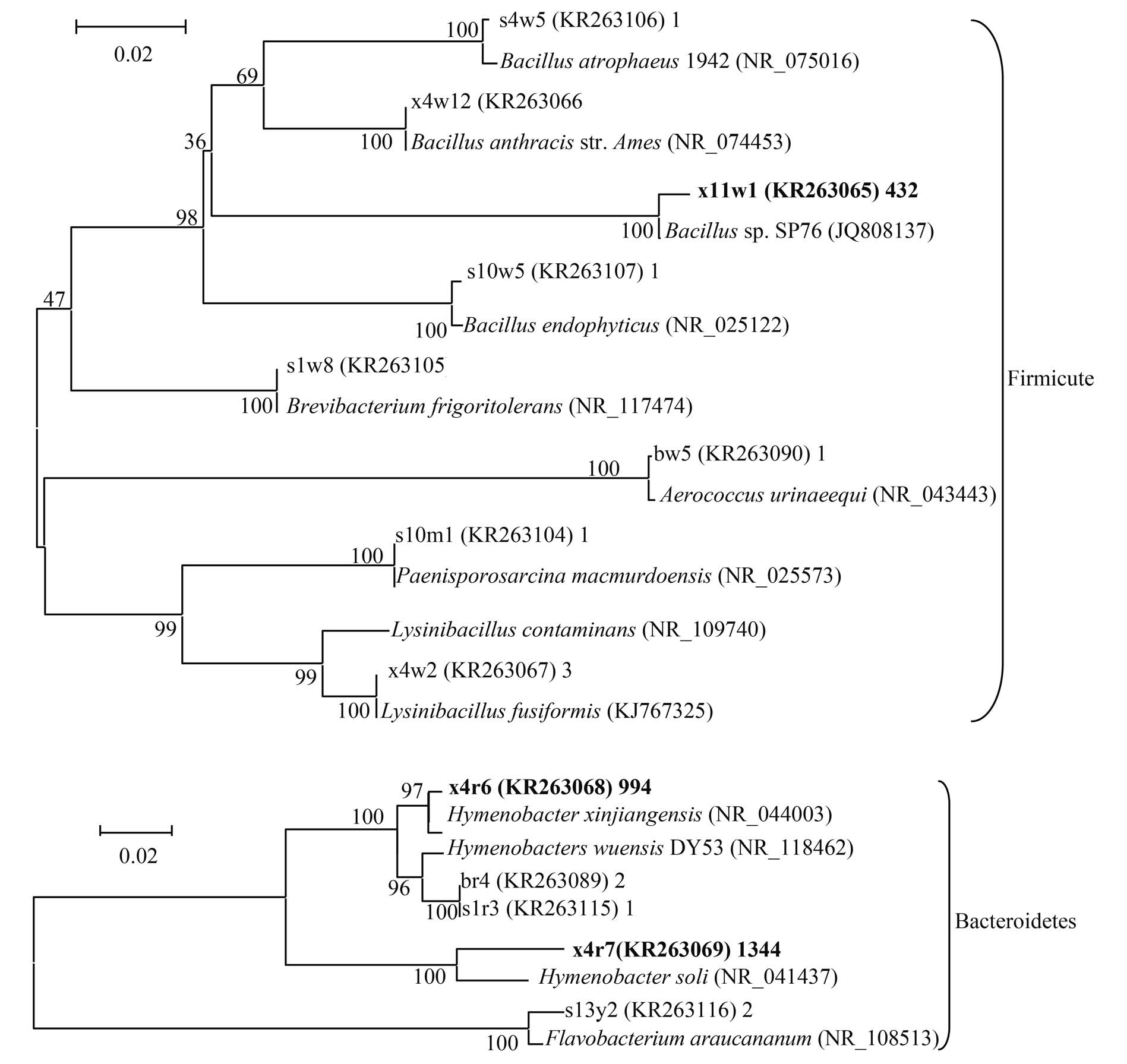
图3.西昆仑崇测冰帽可培养细菌16S rRNA基因序列Firm icutes门和Bacteroidetes门的NJ系统发育树
Figure 3.Neighbor-Joining tree show ing the phylogenetic relationships of culturable bacteria from Chongce Ice Cap based on 16S rRNA gene sequences for Firm icutes and Bacteroidetes.
在上述这些序列中,大部分菌株的序列与GenBank中的序列相似性最高的值在98%-100%,但有7个菌株的序列相似性较低,如:Actinobacteria门的br1与Modestobacter marinus (NR_116228)的序列相似性为96%,bw4与Nocardioides terrigena(NR_044185)的序列相似性为97%,x8a1与Nocardioides sp. TMT2-46-1 (JX949798)的序列相似性为95%。在Proteobacteria门,x4y2和x2w2与Aurantimonas coralicida (NR_042319)的序列相似性分别为96%和97%,s1z1与Massilia p licata (NR_043309)的序列相似性为97%。在Bacteroidetes门,x4r7与Hymenobacter soli (NR_041437)的序列相似性为96%。这7个菌株的序列长度在528-1330 bp,可能是新的属或种,还需做进一步研究。
3 讨论
3.1 不同冰川环境细菌群落结构差异
在属水平上,雪样细菌与冰碛物和土样之间有重合的,但大多数的属是存在差异的。而且,雪样细菌多样性比冰碛物和土样的高。该现象也同时存在于安第斯山脉。该山脉雪样中恢复出8种微生物,而冰碛物中仅恢复出1种微生物[10]。
雪样中的一个优势菌株x4r7不仅与GenBank内的序列相似性比较低,而且该菌株的菌落数为1344,因此在雪样中占据优势(图3)。然而,该菌株在土样和冰碛物中并未恢复出来。这意味着西昆仑崇测冰帽的冰川雪样中可能存在着人类尚未挖掘出的细菌资源。Jacobsen等[11]研究了随着冰川的退缩和消失,冰川流域的淡水生物多样性如何显著的下降。结果表明,不仅是当地的生物多样性比较脆弱,而且灭绝的范围超过了冰川河流已知的稀少特有物种。另外,还有因冰川退缩而引发的冰川径流减少,促进嗜冷的无脊椎动物物种的生活领域逐渐被耐冷的无脊椎动物所侵占,进而向高海拔位移的报道[12-14]。这些研究都说明,气候变化已经或正在对全球的生态系统和生物多样性产生显著影响[15-16],包括使生境退化或丧失,物种灭绝速度加快[17],物种分布范围发生变化[18-20]。如果不对这些资源进行及时的开发和保护,随着冰川退缩的加剧,这些资源很可能在人类尚未意识到的情况下就已经处于濒危状态。
在门的水平上,雪样以Proteobacteria为优势,菌落数为6887;而土样和冰碛物则以Actinobacteria为优势,菌落数分别为169和55(表2)。Proteobacteria的优势状况也同时存在于纽约阿迪朗达克山脉的湖水,以及美国马萨诸塞州的Paker湖等寡营养的环境[21-22]。土样和冰碛物的优势门相同,而与雪样的优势门不同。
在属的水平上,冰川土样中的优势属仅有Arthrobacter,该属在土样和冰碛物中的菌落数分别为168、50,而在雪样中未恢复出来。雪样中的优势属较多,主要有Methylobacterium、Modestobacter、Hymenobacter、Brevundimonas、Bacillus这5种,他们的菌落数分别为6100、2117、994、784、433。虽然前3个在雪样中的菌落数最多,但在土样和冰碛物中没有恢复出来;后面的Brevundimonas、Bacillus这2个属在土样和冰碛物中的菌落数为0-2(表2)。因此,在属的水平上,也是土样和冰碛物的优势属相同,而与雪样的则不同。Liu等[7]对Yala冰川的表层雪、冰碛湖和冰川流的微生物多样性进行了研究。结果为冰碛湖的微生物群落与冰川流的类似,但是雪样中的微生物群落与冰碛湖和冰川流的却不同。在Svalbard冰川,冰尘洞中的真菌群落与邻近的冰碛和苔原土壤的真菌群落差别很大[23]。因此Liu等和Edwards等的研究结果与崇测冰帽的研究结果基本一致。这些研究结果都说明,即使是同一冰川,其不同生境有着非常多样的生态系统。另外,据报道,雪样中的微生物很可能与大气尘埃沉降有关,而尘埃中的微生物群落却与土壤中的微生物群落远远不同[24]。本文中的崇测冰帽正好位于塔克拉玛干沙漠南缘[25],因此其雪样中的微生物很可能来源于该沙漠。这可能也是本研究中雪样中的细菌群落组成与冰碛物和土样的有较大差异的原因。
表2.西昆仑崇测冰帽可培养细菌在属水平上的相对丰度
Table 2.Relative abundance of culturable bacteria in the genus level in Chongce Ice Cap of West KunLun

Phylum Genus Colony number Glacial snow Glacial soil Moraine deposits Actinobacteria Arthrobacter 0 168 50 Kocuria 2 1 0 Modestobacter 2117 0 0 Rhodococcus 0 0 2 Mycobacterium 7 0 0 Nocardioides 2 0 2 Patulibacter 0 0 1 Proteobacteria Pseudomonas 0 3 0 Psychrobacter 2 0 0 Xenophilus 0 17 0 Massilia 0 5 2 Methylobacterium 6100 0 0 Skermanella 0 0 1 Paracoccus 1 0 0 Brevundimonas 784 0 0 Firm icutes Bacillus 433 2 0 Brevibacterium 0 1 0 Aerococcus 0 0 1 Paenisporosarcina 0 1 0 Lysinibacillus 3 0 0 Bacteroidetes Hymenobacter 994 1 2 Flavobacterium 0 2 0
雪样中的优势属M odestobacter、M ethylobacterium和Brevundimonas虽然在土样中未恢复出来,但它们却存在于其它的冰川环境。如Modestobacter在藏色岗日冰川雪[26]中分离到。Methylobacterium是甲烷氧化菌,在天山1号冰川[4]、老虎沟12号冰川[27]等均有报道,Brevundimonas在东绒布冰川[28-29]、Kafni 冰川[30]、老虎沟12号冰川[27]、GISP2[31]也被分离到。上述结果表明,在考虑冰川雪环境细菌菌群的灭绝问题时,应考虑更多的生态系统。
3.2 与其他低温环境相比较
我们将崇测冰帽获得的可培养菌株所在的属与其它冰川位点的属做了比较,如天山1号冰川前沿[4]、北极[32]、东南极岩-水界面[33]、老虎沟12号冰川[27]、东绒布冰川[28-29]、马兰冰川[34-35]、帕隆冰川[2 8]、Ka fn i冰川土样[3 0]、海螺沟冰川[3 6]、GISP2[31]、南极洲Terra Nova湾的水体[37]。比较的结果为,表2中的属,除了Skermanella未在其它冰川环境存在以外,其它的属均有分布。因此该属有可能为崇测冰帽所特有。Skermanella仅分离于冰碛物,说明冰碛物也是开发冰川微生物资源的很好的研究材料。
虽然冰川雪样在融化后可以进入到冰碛物和土样中,但崇测冰帽雪样中的细菌群落在门水平和属水平上,与土样和冰碛物中的差异很大,而土样和冰碛物之间,差异却很小。因此,在冰川退缩的过程中,冰川雪样中的细菌多样性是脆弱的。但在考虑冰川雪环境细菌菌群的灭绝问题时,应考虑更多的生态系统。
参 考 文 献
[1]Xiang SR, Yao TD, An LZ, Wu GJ, Xu BQ, Ma XJ, Li Z, W ang JX, Yu WS. Vertical quantitative and dom inant population distribution of the bacteria isolated from the M uztagata ice core. Science in China (Series D: Earth Sciences), 2005, 48(10): 1728-1739.
[2]Zhang XF, Yao TD, Tian LD, Xu SJ, An LZ. Phylogenetic and physiological diversity of bacteria isolated from puruogangri ice core. Microbial Ecology, 2008, 55(3): 476-488.
[3]Yao TD, Liu YQ, Kang SC, Jiao NZ, Zeng YH, Liu XB, Zhang YJ. Bacteria variabilities in a Tibetan ice core and their relations w ith climate change. Global Biogeochemical Cycles, 2008, 22(4): GB4017, DOI: 10.1029/2007GB003140.
[4]Wu XK, Zhang W, Liu GX, Yang X, Hu P, Chen T, Zhang GS, Li ZQ. Bacterial diversity in the foreland of the Tianshan No. 1 glacier, China. Environmental Research Letters, 2012, 7, DOI:10.1088/1748-9326/7/1/014038.
[5]Liu YQ, Yao TD, Jiao NZ, Kang SC, Huang SJ, Li Q, Wang KJ, Liu XB. Culturable bacteria in glacial meltwater at 6,350 m on the East Rongbuk Glacier, Mount Everest. Extremophiles, 2009, 13(1): 89-99.
[6]Liu YQ, Yao TD, Jiao NZ, Kang SC, Zeng YH, Huang SJ. M icrobial community structure in moraine lakes and glacial meltwaters, Mount Everest. FEMS M icrobiology Letters, 2006, 265(1): 98-105.
[7]Liu YQ, Yao TD, Jiao NZ, Tian LD, Hu AY, Yu WS, Li SH. M icrobial diversity in the snow, a moraine lake and a stream in Himalayan glacier. Extremophiles, 2011, 15(3): 411-421.
[8]Zhang WJ, An RZ, Yang HA, Jiao KQ. Conditions of glacier development and some glacial features in the West Kunlun Mountains. Bulletin of Glacier Research, 1989, 7: 49-58.
[9]Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG. The CLUSTAL_X w indows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25(24): 4876-4882.
[10]González-Toril E, Am ils R, Delmas RJ, Petit J-R, Komárek J, Elster J. Bacterial diversity of autotrophic enriched cultures from remote, glacial Antarctic, A lpine and Andean aerosol, snow and soil samples. Biogeosciences, 2009, 6(1): 33-44.
[11]Jacobsen D, M ilner AM, Brown LE, Dangles O. Biodiversity under threat in glacier-fed river systems. Nature Climate Change, 2012, 2: 361-364.
[12]Brown LE, Hannah DM, M ilner AM. Vulnerability of alpine stream biodiversity to shrinking glaciers and snow packs. Global Change Biology, 2007, 13(5): 958-966.
[13]M ilner AM, Brown LE, Hannah DM. Hydroecological response of river systems to shrinking glaciers. Hydrological Processes, 2009, 23(1): 62-77.
[14]Finn DS, Räsänen K, Robinson CT. Physical and biological changes to a lengthening stream gradient follow ing a decade of rapid glacial recession. Global Change Biology, 2012, 16(12):3314-3326.
[15]Cárdenas ML, Gosling WD, Sherlock SC, Poole I, Pennington RT, Mothes P. The response of vegetation on the Andean Flank in western Amazonia to Pleistocene climate change. Science, 2011, 331(6020): 1055-1058.
[16]Thuiller W, Lavergne S, Roquet C, Boulangeat I, Lafourcade B, Araujo MB. Consequences of climate change on the tree of life in Europe. Nature, 2011, 470(7335): 531-534.
[17]Thomas JA, Telfer MG, Roy DB, Preston CD, Greenwood JJD, Asher J, Fox R, Clarke RT, Law ton JH. Comparative losses of British butterflies, birds, and plants and the global extinction crisis. Science, 2004, 303(5665): 1879-1881.
[18]Colwell RK, Brehm G, Cardelús CL, Gilman AC, Longino JT. Global warm ing, elevational range shifts, and low land biotic attrition in the wet tropics. Science, 2008, 322(5899): 258-261.
[19]Walther GR, Beißner S, Burga CA. Trends in the upward shift of alpine plants. Journal of Vegetation Science, 2005, 16(5):541-548.
[20]W ilson RJ, Gutiérrez D, Gutiérrez J, Martínez D, Agudo R, Monserrat VJ. Changes to the elevational lim its and extent of species ranges associated w ith climate change. Ecology Letters, 2005, 8(11): 1138-1146.
[21]H iorns WD, M ethé BA, Nierzw icki-Bauer SA, Zehr JP. Bacterial diversity in Adirondack mountain lakes as revealed by 16S rRNA gene sequences. Applied and Environmental M icrobiology, 1997, 63(7): 2957-2960.
[22]Zwart G, Crump BC, Kamst-van Agterveld MP, Hagen F, Han SK. Typical freshwater bacteria: an analysis of available 16S rRNA gene sequences from plankton of lakes and rivers. Aquatic M icrobial Ecology, 2002, 28(2): 141-155.
[23]Edwards A, Douglas B, Anesio AM, Rassner SM, Irvine-Fynn TDL, Sattler B, Griffith GW. A distinctive fungal community inhabiting cryoconite holes on glaciers in Svalbard. Fungal Ecology, 2013, 6(2): 168-176.
[24]Stres B, Sul WJ, Murovec B, Tiedje JM. Recently deglaciated high-altitude soils of the Himalaya: diverse environments, heterogenous bacterial communities and long-range dust inputs from the upper troposphere. PLoS One, 2013, 8(9): e76440, DOI: 10.1371/journal.pone.0076440.
[25]Han JK, Liu YX, Liu YJ. Summer temperature trend around Taklimakan Desert reconstructed by the δ18O records of Chongce Ice Core, west Kunlun M ts. Arid Land Geography, 2007, 30(1): 89-93. (in Chinese)韩建康, 刘业祥, 刘永健. 塔克拉玛干沙漠周边地区20世纪前半叶夏季气温趋势变化. 干旱区地理, 2007, 30(1): 89-93.
[26]闫沛迎. 南极与青藏高原冰川雪细菌的多样性研究. 中国科学院大学博士学位论文, 2013.
[27]Zhang S, Hou S, Qin X, Du W, Liang F, Li Z. Prelim inary study on effects of glacial retreat on the dom inant glacial snow bacteria. Geomicrobiology Journal, 2015, 32: 113-118.
[28]Liu Y, Yao T, Jiao N, Kang S, Xu B, Zeng Y, Huang S, Liu X. Bacterial diversity in the snow over Tibetan Plateau Glaciers. Extremophiles, 2009, 13(3): 411-423.
[29]Zhang S, Hou S, Ma X, Qin D, Chen T. Culturable bacteria in Himalayan glacial ice in response to atmospheric circulation. Biogeosciences, 2007, 4(1): 1-9.
[30]Srinivas TNR, Singh SM, Pradhan S, Pratibha MS, Kishore KH, Singh AK, Begum Z, Prabagaran SR, Reddy GSN, Shivaji S. Comparison of bacterial diversity in proglacial soil from Kafni Glacier, Himalayan Mountain ranges, India, w ith the bacterial diversity of other glaciers in the world. Extremophiles, 2011, 15(6): 673-690.
[31]M iteva VI, Sheridan PP, Brenchley JE. Phylogenetic and physiological diversity of m icroorganisms isolated from a deep Greenland glacier ice core. App lied and Environmental M icrobiology, 2004, 70(1): 202-213.
[32]Schütte UME, Abdo Z, Foster J, Ravel J, Bunge J, Solheim B, Forney LJ. Bacterial diversity in a glacier foreland of the high Arctic. Molecular Ecology, 2010, 19 (S1): 54-66.
[33]Huang JP, Swain AK, Thacker RW, Ravindra R, Andersen DT, Bej AK. Bacterial diversity of the rock-water interface in an East Antarctic freshwater ecosystem, Lake Tawani (P). Aquatic Biosystems, 2013, 9: 4, DOI: 10.1186/2046-9063-9-4.
[34]Xiang SR, Yao TD, An LZ, Li Z, Wu GJ, Wang YQ, Xu BQ, Wang JX. Change of bacterial community in the Malan ice core and its relation to climate and environment. Chinese Science Bulletin, 2004, 49(17): 1869-1875.
[35]Xiang SR, Yao TD, An LZ, Xu BQ, Li Z, Wu GJ, Wang YQ, Ma S, Chen XR. Bacterial diversity in Malan ice core from the Tibetan Plateau. Folia Microbiologica, 2004, 49(3): 269-275.
[36]Zhang SH, Yang GL, W ang YT, Hou SG. Abundance and community of snow bacteria from three glaciers in the Tibetan Plateau. Journal of Environmental Sciences, 2010, 22(9):1418-1424.
[37]Giudice AL, Caruso C, Mangano S, Bruni V, de Domenico M, M ichaud L. Marine bacterioplankton diversity and community composition in an Antarctic coastal environment. M icrobial Ecology, 2012, 63(1): 210-223.
(本文责编:张晓丽)
研究报告
Difference of community structure among culturable bacteria in different glacial sam p les on Chongce Ice Cap
Supported by the National Natural Science Foundation of China (31100369, 41101072, 41330526)*Corresponding author. Tel: +86-370-3115311; E-mail: shuhongzhang_2013@163.com
Received: 12 August 2015; Revised: 13 September 2015; Published online: 22 October 2015
Abstract: [Objective] We studied the difference of bacterial community composition among glacial snow, moraine deposits and glacial soil on Chongce Ice Cap of West Kunlun M ountains. [M ethods] Based on traditional culturedependent and 16S rRNA sequence analysis, we analyzed the community structure of bacteria on the level of genus and phylum. [Results] Results show that glacial bacteria were composed of Firm icutes, Proteobacteria, Actinobacteria and Bacteroidetes on the phylum level. Glacial snow was dom inated by Proteobacteria, whereas glacial soil and moraine deposits were dom inated by Actinobacteria. On the genus level, glacial soil was dom inated by Arthrobacter, while glacial snow w as dom inated by Methylobacterium, Modestobacter, Hymenobacter, Brevundimonas and Bacillus. Bacterial composition was sim ilar between glacial soil and moraine deposits, but different from glacial snow. Skermanella may be unique on Chongce Ice Cap. [Conclusion] Our study indicated the vulnerability of bacterial diversity in glacial snow w ith glacial retreat, and the importance of bacterial resources preservation on glacial snow environments.
Keywords: culturable bacteria, community structure, Chongce Ice Cap, glacier
基金项目:国家自然科学基金(31100369,41101072,41330526)
收稿日期:2015-08-12;修回日期:2015-09-13;网络出版日期:2015-10-22
