天山一号冰川底部沉积层产蛋白酶耐低温菌株的筛选及其系统发育
【类型】期刊
【作者】倪永清,顾燕玲,史学伟,郑晓吉,韩亮,周红,程国栋(中国科学院寒区旱区环境与工程研究所冻土工程国家重点实验室;石河子大学食品学院)
【作者单位】中国科学院寒区旱区环境与工程研究所,冻土工程国家重点实验室;石河子大学食品学院
【刊名】微生物学报
【关键词】 天山冰川;耐低温菌株;蛋白酶;系统发育
【资助项】国家自然科学基金项目 (40961002;41140009);冻土工程国家重点实验室开放基金项目 (SKLFSE200903);中国博士后科学基金特别资助 (20100339...
【ISSN号】0001-6209
【页码】P164-172
【年份】2019
【期号】第2期
【期刊卷】1;|7;|8;|4;|5;|2;|12
【摘要】【目的】通过对天山一号冰川底部沉积层耐低温菌的分离和其中产蛋白酶菌株的筛选,了解冰川微生物生理多样性和系统发育多样性,为高效低温蛋白酶生物技术的研发奠定基础。【方法】采用稀浓度的R2A、TSB平板涂布分离可培养细菌,通过脱脂乳选择性培养基筛选产蛋白酶的耐低温菌株。对分离菌株表型特征、生理生化特性、最适生长温度、耐盐性、产酶性能进行了比较,结合16S rRNA基因序列同源性分析确定产蛋白酶菌株的多样性和系统进化地位,通过BOX-PCR指纹技术分析16S rRNA基因序列高相似度的近缘菌株的遗传差异。【结果】从125株分离物中筛选到27株产蛋白酶的耐低温菌株,其中21株为适冷菌,仅6株菌为专性嗜冷菌,革兰氏阴性菌居多,假单胞菌属(Pseudomonas)菌株占40.7%。产酶菌株隶属于5个系统发育类群、9个属,其中γ-Proteobacteria、Actinobacteria、CFB(Cytophaga-Flexibacter-Bacteroides)为优势类群。【结论】天山1号冰川底部沉积层冻土中产蛋白酶的耐低温细菌多样性较丰富,本研究筛选得到的同属近缘种群较多,其产酶性状存在差异,适合开展微生物种群的生物地理学研究。
【全文】
天山一号冰川底部沉积层产蛋白酶耐低温菌株的筛选及其系统发育
摘要:【目的】通过对天山一号冰川底部沉积层耐低温菌的分离和其中产蛋白酶菌株的筛选,了解冰川微生物生理多样性和系统发育多样性,为高效低温蛋白酶生物技术的研发奠定基础。【方法】采用稀浓度的R2A、TSB平板涂布分离可培养细菌,通过脱脂乳选择性培养基筛选产蛋白酶的耐低温菌株。对分离菌株表型特征、生理生化特性、最适生长温度、耐盐性、产酶性能进行了比较,结合16S rRNA基因序列同源性分析确定产蛋白酶菌株的多样性和系统进化地位,通过BOX-PCR指纹技术分析16S rRNA基因序列高相似度的近缘菌株的遗传差异。【结果】从125株分离物中筛选到27株产蛋白酶的耐低温菌株,其中21株为适冷菌,仅6株菌为专性嗜冷菌,革兰氏阴性菌居多,假单胞菌属(Pseudomonas)菌株占40.7%。产酶菌株隶属于5个系统发育类群、9个属,其中 γ-Proteobacteria、Actinobacteria、CFB(Cytophaga-Flexibacter-Bacteroides)为优势类群。【结论】天山1号冰川底部沉积层冻土中产蛋白酶的耐低温细菌多样性较丰富,本研究筛选得到的同属近缘种群较多,其产酶性状存在差异,适合开展微生物种群的生物地理学研究。
关键词:天山冰川,耐低温菌株,蛋白酶,系统发育
地球生物圈四分之三属于永久低温环境,包括南北两极及环极地地区、深海海水、高原高山地区[1-2]。这些生态环境中生存着大量微生物,根据最适生长温度大体可分两类,最适生长温度小于等于15℃、最高生长温度小于20℃,能够在0℃甚至更低温度生长的称为嗜冷微生物;相反能够在0℃生长,但最适生长温度在24℃左右,最高生长温度在30-37℃的称为耐冷微生物。来自低温环境的微生物从大类群上包括细菌、酵母菌、霉菌、微藻等。由于生理上独特的适应性和大分子结构方面特殊性(包括细胞膜脂质的特殊性、酶分子结构的柔性以及冷激蛋白、低温保护剂分子的快速合成),这些微生物能够在低温环境中生存、繁衍[1-2]。这些微生物分泌的胞外酶在低温下和中度温度下有较高的催化活性,在温度适当提高后就可以使这些低温酶失活,因此这些酶不仅在低温环境有机质的循环中发挥着重要的生态学作用,在农业、能源、食品、医药、纺织、环境治理等领域也有广泛的应用价值,目前从低温微生物中筛选新的生物活性物质已成为新的研究热点[3-4]。
蛋白酶(Protease)是水解蛋白质的一大类酶的通称,广泛应用于食品、洗涤添加剂、饲料、造纸、制药等行业,占世界用酶市场60%以上的份额。低温蛋白酶是一个相对概念,其最适作用温度通常比同功能的中温(50℃)蛋白酶要低20-30℃左右,且在较低的温度下(0-20℃)拥有较高的酶活性,而在高温条件下(>60℃)的酶活性较差[5]。正是由于这些特点使得低温蛋白酶在食品加工业、冷水洗涤剂、动植物抗冻、医用疫苗以及生物保鲜方面有着普通中高温蛋白酶无法取代的优越性。例如在肉类工业中,低温蛋白酶可以在低温条件下作用结缔组织胶原和弹性硬蛋白,在50℃左右失活,即使在pH值4-5的条件下也有很好的活性,所以是理想的肉类柔嫩酶制剂[6]。
自20世纪70年代以来,相继报道了低温蛋白酶的研究,已从海冰、嗜冷的鱼类和贝类、南北极及环北极的高纬度冻土、高海拔高山冰川、冻土等环境中分离到产低温蛋白酶的菌株[3]。国内目前对于产低温蛋白酶菌株的研究大多来自南极及南海深部沉积层,而对分布广泛的高海拔低温蛋白酶菌株的研究报道较少[7-10]。天山山脉横亘新疆中部约1700公里,海拔4000多米的山峰终年积雪,形成永久冻土带和冰川,为低温微生物资源的开发提供了丰富的来源。本文对天山一号底部冰、水、岩沉积层永久冷冻环境中产低温蛋白酶的菌株进行了筛选、纯化,并研究了其系统发育多样性,以期为低温蛋白酶生物技术的研发奠定基础。
1 材料和方法
1.1 材料
1.1.1 主要试剂和仪器:用于 PCR扩增的全套试剂及扩增引物均购自 TaKaRa公司。相关生理生化试验所用试剂均购自于天津市巴斯夫化学试剂厂及天津市致远化学试剂厂。高速冷冻离心机为Thermo公司Fresco21型;PCR仪为德国Biometra公司Tprofessional;凝胶成像系统为 BioRad公司 Gel DOC XR;水平电泳仪为美国 BioRad公司PowerPac Universal、电泳槽 SUBCELL GT(20 ×25 cm)。
1.1.2 培养基:耐低温菌株的分离和培养根据Shi等人的方法进行[11],产蛋白酶菌株的筛选采用1/2×TSB+2.5%脱脂乳培养基。
1.2 样品采集
样品于2010年7月22日、2011年9月10日采集自天山一号冰川西支尾部的底部沉积层,海拔为3833 m(43°07.125N,86°48.707E)。将冰、水、岩混合沉积层样品迅速装入己灭菌的保鲜盒内,置于车载冰箱中-4℃保存,12小时之内运回实验室,部分样品保存在-70℃超低温冰箱;按样品、蒸馏水1∶3的比例在摇床震荡10 min后静置30 min,用标准pH计测定土壤浸出液pH值。以上所有过程均在无菌条件下完成。
1.3 菌株的分离与纯化
在超净工作台上削去表层可能受到污染的样品,采用梯度稀释液直接平板涂布法对土壤样品进行分离,10℃、18℃分别培养5-10 d。连续转接划线培养3次后,根据菌落颜色、大小、形态等表型差异进行初步分离筛选并纯化,所得纯培养物转接TSB斜面培养基4℃保藏备用。液体纯培养物离心后,加入新鲜培养液重悬,补充15%的灭菌甘油冷冻包藏在-70℃冰箱。
1.4 产低温蛋白酶菌株筛选
经液体培养基增菌后,选择适宜的稀释菌液划线于1/4×R2A+2.5%脱脂乳培养基平板上,以菌落透明圈作为初筛依据,选择透明圈较大、菌落直径较大的菌株于斜面培养基4℃保存。
1.5 产酶菌株生长特性
菌株最适生长温度测定:将产酶菌株的液体培养液按0.5 mL的接入量接入装有5 mL液体富集培养基的试管中,分别置于 4℃、10℃、15℃、18℃、24℃、30℃、37℃ 七个温度梯度下培养,培养24 h后420 nm测OD值。
菌株的耐盐性测定:在液体培养基中分别加入1% 、4% 、6% 、8% 、10% 、12% 、14% 、16% 浓度的氯化钠溶液,接入产酶菌株,在12℃下培养一周后,420 nm测OD值。
1.6 16S rRNA基因PCR扩增和系统进化分析
参照林万明等人的方法[12]提取单菌落的DNA,采用细菌 16S rRNA基因通用引物27f、1492r进行 PCR 扩 增。PCR 产 物 用 AxyPrepTMPCR Cleanup Kit(AxyGEN)试剂盒纯化后,由上海美吉生物科技有限公司采用ABI377DNA自动测序仪上直接测序。将测序结果提交到GenBank数据库中,用BLAST进行相关序列的搜索[13],获得相关种、属的16SrDNA序列。CLUSTAL X 1.83软件序列联配[14],进化距离采用邻接法 neighbor-joining法,依据p-distances法和 Kimura-2parameter双参数法,用MEGA v.5.0 软件建立系统发育树[15],进化树分支的置信度采用bootstrap法,重复次数为1000。
1.7 产酶菌株rep-PCR指纹图谱分析
采用单引物BOXAIR(5-CTA CGG CAA GGC GAC GCT GAC G-3)进行 PCR 扩增[16],取 10μL 扩增产物在1.5%琼脂糖凝胶、0.5×TBE电泳缓冲液、4V/cm条件下电泳检测2 h。根据 PCR扩增带谱的有无转化为含有1、0两值变量的矩阵,采用Jaccard相似性系数,使用 NTSYS-pc 2.01(Applied Biostatistics,Inc)软件采用非加权算术平均连锁法(UPGMA,unweighted pair group method with arithmetic mean)进行聚类分析。
1.8 产酶菌株生理生化特征
参照文献[17]进行。
2 结果
2.1 产酶菌株的筛选
纯化后的菌株依据菌落形态、颜色、细胞形态、革兰氏染色与液体培养特征观察,根据产蛋白酶鉴别培养基上是否出现蛋白水解圈,从冰川底部沉积层样品中分离的125个菌株中共筛选得到58株疑似产蛋白酶的可培养物,其中27株产酶性能良好。革兰氏染色结果表明,27株产低温蛋白酶菌株中有17株为革兰氏阴性菌,占总数的63%;10株为革兰氏阳性菌,占总数的37%。产酶菌株的产酶情况、菌落颜色、大小、形态、细胞形态见表1。a+++ ,diameter ratio≥3;++ ,diameter ratio≥2;+:diameter ratio≥1.bThe optimal growth temperature.
表1 天山1号冰川底部沉积层产蛋白酶低温菌株的特征
Table 1 Characteristics of cold-adapted bacterial strains producing protease from permafrost sediments of the bottom layer of the Glacier No.1 in the Tianshan Mountains
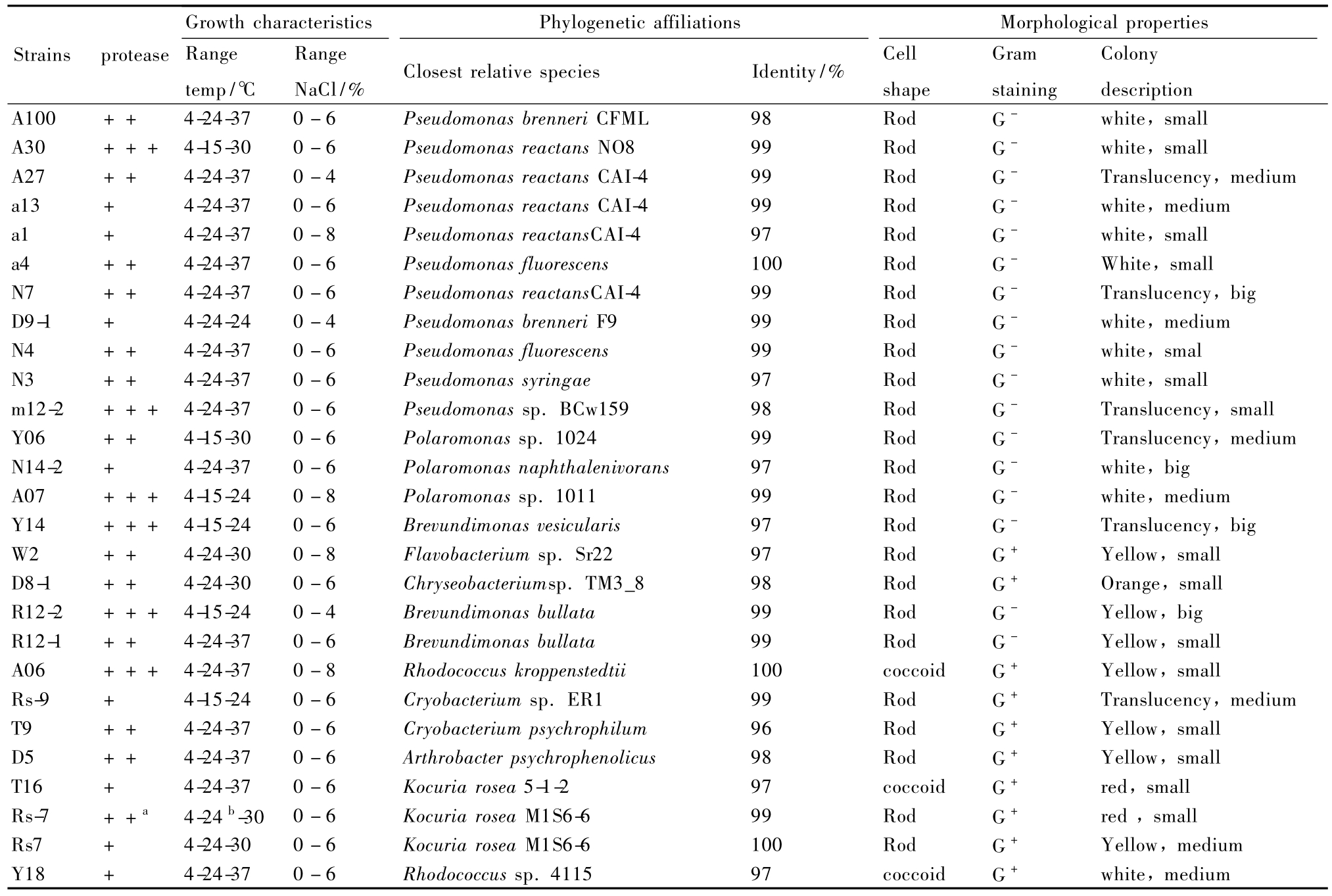
Growth characteristics Phylogenetic affili Strains protease Range Colony description A100 ++ 4-24-37 0-6 Pseudomonas brenneri CFML 98 Rod G- white,ations Morphological properties Range temp/℃NaCl/% Closest relative species Identity/% Cell shape Gram staining medium small A30 +++ 4-15-30 0-6 Pseudomonas reactans NO8 99 Rod G- white,small A27 ++ 4-24-37 0-4 Pseudomonas reactans CAI-4 99 Rod G- Translucency,medium a13 + 4-24-37 0-6 Pseudomonas reactans CAI-4 99 Rod G- white,medium a1 + 4-24-37 0-8 Pseudomonas reactansCAI-4 97 Rod G- white,small a4 ++ 4-24-37 0-6 Pseudomonas fluorescens 100 Rod G- White,small N7 ++ 4-24-37 0-6 Pseudomonas reactansCAI-4 99 Rod G- Translucency,big D9-1 + 4-24-24 0-4 Pseudomonas brenneri F9 99 Rod G- white,medium N4 ++ 4-24-37 0-6 Pseudomonas fluorescens 99 Rod G- white,smal N3 ++ 4-24-37 0-6 Pseudomonas syringae 97 Rod G- white,small m12-2 +++ 4-24-37 0-6 Pseudomonas sp.BCw159 98 Rod G- Translucency,small Y06 ++ 4-15-30 0-6 Polaromonas sp.1024 99 Rod G- Translucency,medium N14-2 + 4-24-37 0-6 Polaromonas naphthalenivorans 97 Rod G- white,big A07 +++ 4-15-24 0-8 Polaromonas sp.1011 99 Rod G- white,medium Y14 +++ 4-15-24 0-6 Brevundimonas vesicularis 97 Rod G- Translucency,big W2 ++ 4-24-30 0-8 Flavobacterium sp.Sr22 97 Rod G+ Yellow,small D8-1 ++ 4-24-30 0-6 Chryseobacteriumsp.TM3_8 98 Rod G+ Orange,small R12-2 +++ 4-15-24 0-4 Brevundimonas bullata 99 Rod G- Yellow,big R12-1 ++ 4-24-37 0-6 Brevundimonas bullata 99 Rod G- Yellow,small A06 +++ 4-24-37 0-8 Rhodococcus kroppenstedtii 100 coccoid G+ Yellow,small Rs-9 + 4-15-24 0-6 Cryobacterium sp.ER1 99 Rod G+ Translucency,medium T9 ++ 4-24-37 0-6 Cryobacterium psychrophilum 96 Rod G+ Yellow,small D5 ++ 4-24-37 0-6 Arthrobacter psychrophenolicus 98 Rod G+ Yellow,small T16 + 4-24-37 0-6 Kocuria rosea 5-1-2 97 coccoid G+ red,small Rs-7 ++a 4-24b-30 0-6 Kocuria rosea M1S6-6 99 Rod G+ red,small Rs7 + 4-24-30 0-6 Kocuria rosea M1S6-6 100 Rod G+ Yellow,medium Y18 + 4-24-37 0 -6 Rhodococcus sp.4115 97 coccoid G+ white,
2.2 产酶菌株最适生长温度与耐盐性
实验结果表明(表1),27株产酶菌株中21株菌最适生长温度为24℃左右,多数菌株在37℃即停止生长,属耐冷菌;只有6株菌最适生长温度为15℃左右,生长温度范围在0℃ -24℃,属于专性嗜冷菌。在耐盐性研究中,产酶菌株大多数最大耐盐度为6%,耐盐性最高的菌株是m12-2,在8%时仍可以生长,但生长缓慢,而其它大多数菌株在此浓度下停止生长。
2.3 基于16S rRNA基因序列的产酶菌株系统发育
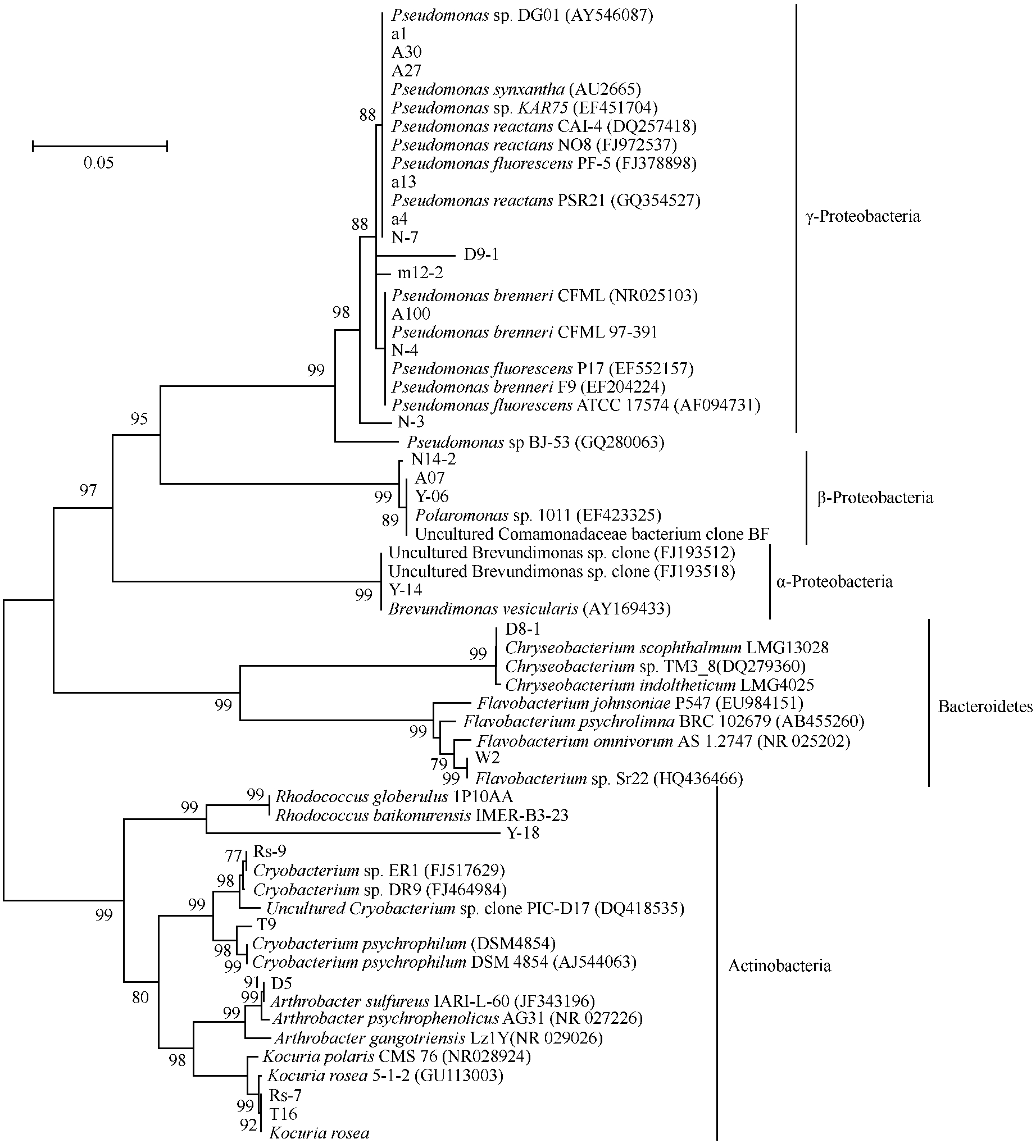
图1 基于部分16S rRNA基因序列的产蛋白酶低温菌株系统发育树
Fig.1 Neighbour-joining tree showing the phylogenetic relationships among protease-producing strains 16S rRNA gene partial sequences and their closely related sequences downloaded from GenBank.Numbers at the nodes indicate the bootstrap values(70%)based on neighbour-joining analyses of 1000 resampled datasets.Bar:5%sequence divergence.
从天山一号冰川尾部沉积层含冰冻土样品中分离到细菌共125株,筛选到产低温蛋白酶的菌株27株,图1为构建的系统发育树。由系统发育树可知27株产蛋白酶菌株分属于 α变形细菌亚纲(α-Proteobacteria)、β 变形细菌亚纲(β-Proteobacteria)、γ变形细菌亚纲(γ-Proteobacteria)、放线菌门(Actinobacteria)、CFB (Cytophaga-Flexibacter-Bacteroides)5个门(或亚纲)水平的系统发育类群。图1和表1显示,产低温蛋白酶的菌株隶属于γ变形细菌亚纲的最多,共11株,占全部分离菌株总数的40.7%,根据16S rRNA基因序列相似性,全部归属于 Pseudomonas属,其中菌株 A30、A27、a13、a1、N7与 Pseudomonas reactan亲缘关系较近,菌株A100、D9-1与 Pseudomonas brenneri亲缘关系较近,菌株N3与Pseudomonas syringae亲缘关系最近,a4、N4两株菌与Pseudomonas fluorescens等多株典型菌株16S rRNA基因序列相似性在99%以上。此外,菌株 N14-2、A07、Y06属于 β 变形细菌亚纲的Polaromonas属,其 中 N14-2 与 Polaromonas naphthalenivorans的相似性达到 97%,而 A07、Y06与 Polaromonas hydrogenivorans、Polaromonas aquatica等多株典型菌株16S rRNA基因相似性在98%以上。隶属于α变形细菌亚纲的菌株包括Y14、R12-2、R12-1,均归属于 Brevundimonas属,菌株 Y14 与Brevundimonas vesicularis具有97%的相似性,后两株菌与Brevundimonas bullata具有99%的序列相似性。在筛选的产蛋白酶菌株中,虽然隶属于放线菌门(Actinobacteria)的菌株只有 7株,但依据 16S rRNA基因序列相似性标准,表现为最高的多样性,分属于4个属,其中菌株 Y18、A06为 Rholococces属成员;菌株 Rs-9、T9隶属于 Cryobacterium属,其中T9与Cryobacterium psychrophilum DSM 4854具有98%的序列相似性;菌株 Rs-7、T16隶属于 Kocuria属,与菌株 Kocuria rosea M1S6-6的序列相似性达98%;菌株D5与节杆菌 Arthrobacter psychrophenolicus具有98%的序列相似性。隶属于CFB门的细菌菌株包括D8-1和 W2,分别与可培养菌株 Chryseobacterium sp.TM3_8、Flavobacterium sp.Sr22具有98%、97%的16S rRNA基因序列同源性。
2.4 产酶菌株rep-PCR指纹聚类分析
研究分离到的产低温蛋白酶菌株最多的是隶属于γ变形细菌亚纲的 Pseudomonas,共11株,16S rRNA基因序列分析表明菌株之间的同源性在97%以上,这些菌株与多个典型的 Pseudomonas种的同源性均在98%以上,不能准确确定它们的系统发育和遗传差异。采用BOX-PCR指纹图谱显示了Pseudomonas属内菌株以及其它系统发育类群菌株在基因组水平上的差异(图2A)。可以看出每个菌株产生的条带数在5-11之间,图2B为指纹图谱聚类分析的树状图,其中 T9、D8-1、W2、Y14四株菌与Pseudomonas不同门不同属,表现出最大的遗传差异;11株Pseudomonas spp.菌株在85%的相似性水平上,可划分为四个群,其中 N3、N4、A100与 A30为一群,A27与a13、a4与N7各自成一群,具有完全相同的带谱,应该分别对应于三个不同的种;D9-1与m12-2指纹图稍有差别,可能对应于属内另外一个亲缘关系较远的Pseudomonas种。例外的是菌株Y06,本属于β-Proteobacteria亚纲细菌,但指纹图谱显示与Pseudomonas遗传关系较近。

图2 产蛋白酶菌株的BOX-PCR指纹图谱及聚类分析树状图
Fig.2 BOX-PCR fingerprint patterns of isolates in 1.5%agarose(A).Lanes:1,W2;2,a13;3,A27;4,N3;5,A100;6,N4;7,a4;8,A30;9,N7;10,Y14;11,a1;12,T9;13,Y06;14,N14-2;15,D8-1;16,m12-2;17,a4;18,D9-10;C,blank;M,mark 4000bp ladder.Dendrogram obtained by UPGMA cluster analysis of the BOX-PCR datasets(B).
2.5 产酶菌株典型生理生化特征
表2为细菌常规生理生化实验结果,结果表明假单胞菌属(Pseudomonas)的产酶菌株接触酶阳性,大部分明胶水解呈阴性,多数V-P实验阳性,大部分硝酸盐还原实验阴性,菌株之间生理差异与指纹图谱聚类分群结果基本一致。D9-1、T16、A06同属于 Rhodococcus,明胶水解阴性,M.R.、V-P、接触酶阳性,硝酸盐还原阳性;Y6与N14-2生理特性相同,R12-1、R12-2、Rs-9、T9、D5、Rs7、Rs-7 同 属Actinobacteria,但代表不同的种属,生理生化特征也有差异,多数明胶水解、硝酸盐还原阴性,M.R.、接触酶实验阳性,V-P实验阴性。
表2 产低温蛋白酶菌株的生理生化特征
Table 2 Physiological and biochemical characteristics of strains producingcold-active protease
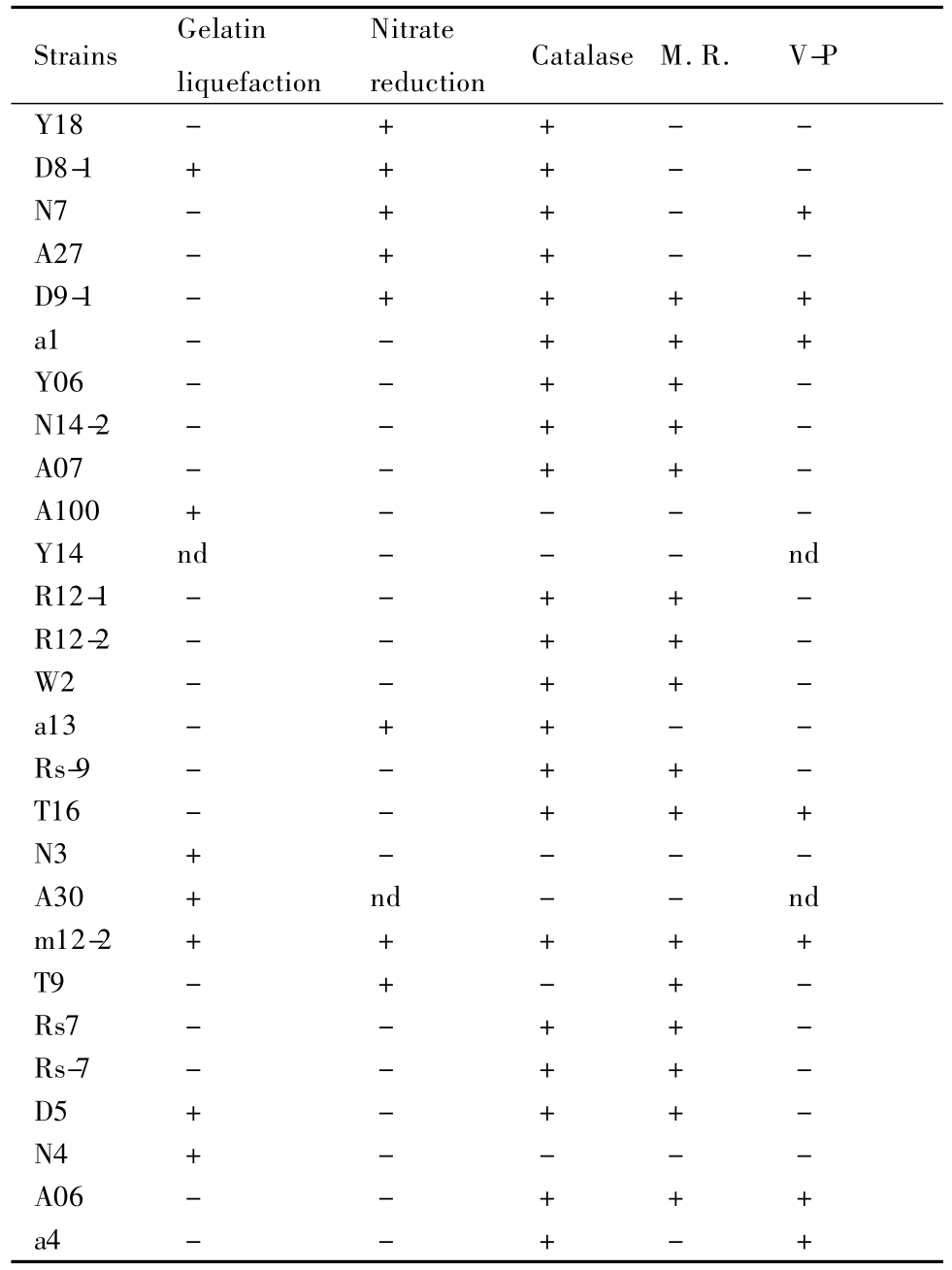
-,negative;+,positive;nd,not determined.
Strains Gelatin liquefaction Nitrate reduction CatalaseM.R. V-P Y18- + +- -D8-1+ + +- -N7- + +-+A27- + +- -D9-1 - + + + +a1- -+++Y06- - ++-N14-2- - ++-A07- - ++-A100+ - - - -Y14nd - - -nd R12-1- - ++-R12-2- - ++-W2- - ++-a13- + +- -Rs-9- - ++-T16- - +++N3+ - - - -A30+ nd - -nd m12-2 + + + + +T9- + -+-Rs7- - ++-Rs-7- - ++-D5+ -++-N4+ - - - -A06- - +++a4- - +-+
3 讨论
低温蛋白酶在食品加工业、冷水洗涤剂、动植物抗冻、制药、医用疫苗以及生物保鲜方面极具应用潜力和商业价值。虽然地球生物圈80%以上为低温环境,包括两极及近极地区域、深海、海冰、高山冰川冻土,为产低温酶菌株资源的开发应用提供了极其丰富的源泉[3]。但是到目前为止,低温酶生物技术的应用开发并不成熟,在资源微生物挖掘的广度上和低温酶作用机制、遗传工程改进的深度方面需要开展更深入的研究[3,6]。
根据文献报道,低温环境中分离的细菌大多数属于耐冷细菌,最适生长温度在20-25℃,只有部分菌株为专性嗜冷菌,最适生长温度在10-15℃。从系统发育来看,无论海冰、还是冰川冻土等低温环境中,目前可培养的细菌包括6个系统发育类群:α-Proteobacteria、β-Proteobacteria、γ-Proteobacteria、Actinobacteria、CFB( Cytophaga-Flexibacter-Bacteroides)、Firmicutes,前五个是低温环境中最主要的系统发育类群,其中至少包括35个以上的属[1,2,18]。但有证据表明,不同气候、不同地理基岩环境的冰川、冻土中,微生物群落在不同层次分类单元上的组成、丰度明显不同[19]。最近针对高山冻土的分子生物学方法研究显示,Proteobacteria丰度占据绝对优势,其中β-Proteobacteria是最具优势的类群,但是采用分离培养物的研究方法得到的结果并不一致,冻土中革兰氏阳性菌和革兰氏阴性菌数量相当,其中 γ-Proteobacteria占据了可培养分离物的优势类群[20]。在本研究中,采用可培养方法结果显示,Proteobacteria和 Actinobacteria门细菌丰度占优势,其中隶属于γ-Proteobacteria的Pseudomonas可培养物数量最多,其次为 Actinobacteria门的Arthrobacter、Rhodococcus,而隶属于 Firmicutes的菌株数量很少,只分离到一株与 Paenibacillus polymyxa 16S基因序列同源性在98%的菌株,另外一株属于Enterococcus durans(本文中未报道),但都未显示出具有产蛋白酶的特性。
目前发现,低温环境中分离的产蛋白酶的菌株,最常见的是隶属于γ-Proteobacteria亚纲的以下几个细 菌属: Pseudoalteromona、Pseudomonas、Psychrobacter、Stenotrophomonas、Shewanella、Colwellia,以及隶属于CFB的细菌Sphingobacterium、Flavobacterium,而Actinobacteria中最常见的是Arthrobacter、Rhodococcus属,相比较而言隶属于Firmicutes的菌株很少,其中 γ-Proteobacteria亚纲的Shewanella、Colwellia 主要发现于深海沉 积层[21]。本研究结果显示,天山一号冰川底部沉积层中产蛋白酶的菌株中丰度最高的是Pseudomonas和Arthrobacter,系统发育分析显示,Pseudomonas为多样性最高的分类单元(图1和2)。此外,从天山一号冰川底部沉积层中分离的产蛋白酶菌株丰富较高的还有 β-Proteobacteria亚纲的 Polaromonas属,而α-Proteobacteria亚纲的菌株很少。与可培养的 γ-Proteobacteria细菌菌株的种数相比,本研究中得到β-Proteobacteria亚纲的可培养物种数比例明显偏低。但是我们开展的高通量测序技术研究显示,丰度和多样性统计均表明一号冰川底部沉积层中β-Proteobacteria、CFB门细菌占绝对优势,分别包括157、141个OTU(97%的相似性水平统计)、分别占总的测序reads数的52.86%、26%(另撰文报道),而γ-Proteobacteria亚纲的序列只占3.35%。这可能是由于本研究采用的培养方法和产酶底物脱脂牛奶可能更有利于 γ-Proteobacteria的培养和筛选,不利于其它类群的分离[22]。尤其在一号冰川底部沉积层中,测序统计的隶属于Acidobacteria的reads占总数的0.68%。但是到目前为止,所有冷冻低温环境中分离的菌株,隶属于 Acidobacteria门的可培养细菌几乎为零。这其中的原因,一是可能由于到目前为止人们采用的培养基和底物对微生物的选择性培养,另一方面是微生物在营养成分的利用上很可能形成了复杂的代谢网络,其生长条件互为因果[23]。尤其是冰川及其底部沉积层,由于表层融水的不断渗透溶解,有机质养分不能得到补充,相比其它冻土环境,这样的极端自然环境有机质营养更加贫瘠,分离的微生物均属于寡营养型,在普通培养基的富营养状态下难以生长。因此,针对这些环境中的微生物资源开发,一方面需要进一步在培养方法和技术上取得突破,另一方面需要开展宏基因组学方法挖掘功能酶及其基因资源。
本研究中冰川沉积层分离得到的耐低温的Pseudomonas、Arthrobacter、Rhodococcus、Polaromonas菌株,其16S rRNA基因序列与分类学地位明确的多个同属细菌典型菌株的相似性在98%以上,甚至99%以上,因此鉴定最多只能划分在属(genus)一级水平[24]。此外,虽然本研究分离的同属细菌菌株相互之间的16S rRNA基因序列同源性也在97-98%以上,但它们的菌落形态、颜色等表型特征以及产酶情况有差异(包括不产酶、产其它酶性状)[25-26],这说明依据16S rRNA基因序列差异标准不能准确反映冰川等冰冻圈环境中的优势种群的系统发育和多样性,需要采用更精确的分子手段确定,这个结果与Susana等人从南极分离的产蛋白酶的Pseudomonas种群相似[27]。采用基于全基因组的 rep-PCR指纹技术和生理学代谢谱可以将16S rRNA基因序列同源性很高的许多Pseudomonas菌株区分为不同的亚群,可对应于同属不同的种或者种组[28]。同理,微生物菌株的产酶性状谱也应该反映种群内的组群特征,显示了微生物种群或者亚种群趋异分化的代谢组学特征,这与目前采用代谢组学技术手段鉴定微生物、揭示微生物种群进化的方法相一致[29]。因此,针对特殊的低温和寡营养环境,以不同环境来源的、16S rRNA基因序列亲缘关系很近的菌株作为研究对象,在种群水平上开展微生物遗传分化、微进化、成种机制以及微生物地理学的研究将极具科学意义和价值。
参考文献
[1]Rodrigues DF,Tiedje JM.Coping with our cold planet.Applied and Environmental Microbiology,2008,74(22):1677-1686.
[2]Margesin R, Miteva V. Diversity and ecology of psychrophilic microorganisms.Research in Microbiology,2011,162(3):346-361.
[3]Huston AL.BiotechnologicalAspectsofcold-adapted enzymes//Margesin R,Schinner F,Marx JC,Gerday C.Psychrophiles:from biodiversity to biotechnology.Berlin Heidelberg:Springer,2008:347-364.
[4]Gerday CM,Aittaleb M,Bentahir JP,Chessa P,Claverie T,Collins S,D’Amico.Cold-adapted enzymes:from fundamentalsto biotechnology. TrendsBiotechnology,2000,18(3):103-107.
[5]Dastager SGA,Dayanand WJ.Li CJ,Kim JC,Lee DJ,Park XP,Tian,Raziuddin QS.Proteolytic activity from an alkali-thermotolerant Streptomyces gulbargensis sp.nov.Current Microbiology,2008,57(6):638-642.
[6]Feller G,Gerday C.Psychrophilic enzymes:hot topics in cold adaptation.Nature Review Microbiology,2003,1(3):200-208.
[7]Zhang R,Zen R.Isolation of deep-sea strain that produce alkaline protease and studies on ITS fermentation condition.Microbiology China,2001,28(4):5-9.(in Chinese)张锐,曾润颖.极端微生物产碱性蛋白酶菌株的筛选及发酵条件的研究.微生物学通报,2001,28(4):5-9.
[8]Zeng Y,Cai M,Chen B,He J.Characterization of an arctic marine bacterium producing protease.Chinese Journal of Polar Research,2000,12(4):263-268.(in Chinese)曾胤新,蔡明红,陈波,何剑锋.一株北极海洋细菌及其蛋白酶的生理生化特征研究.极地研究,2000,12(4):263-268.
[9]Zeng R,Zhang R,Zhao J,Lin N.Cold-active serine alkaline protease from the psychrophilic bacterium Pseudomonasstrain DY-A: enzyme purification and characterization.Extremophiles,2003,7(4):335-337.
[10]Zhou MY,Chen XL,Zhao HL,Dang HY,Luan XW,Zhang XY,He HL,Zhou BC,and Zhang YZ.Diversity of both the cultivable protease-producing bacteria and their extracellular proteases in the sediments of the South China Sea.Microbial Ecology,2005,58(3):582-590.
[11]Shi T,Reeves RH,Gilichinsky DA,Friedmann EI.Characterization of viable bacteria from Siberian permafrost by 16S rDNA sequencing.Microbial Ecology,1997,33(3):169-179.
[12]林万明.《细菌分子遗传学分类鉴定法》.上海:上海科学技术出版社,1990.
[13]Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees.Molecular Biology Evolution,1987,(4):406-425.
[14]Thompson JD,Gibson TJ,Plewniak F,Jeanmougin F,Higgins DG.The CLUSTAL X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools.Nucleic Acids Research,1997,25(24):4876-4882.
[15]Tamura K,Peterson D,Peterson N,Stecher G,Nei M,and Kumar S.MEGA5:Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance,and Maximum Parsimony Methods.Molecular Biology and Evolution,2011,28(10):2731-2739.
[16]VersalovicJ, SchneiderM, deBruijn FJ. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction.Methods in Molecular and Cellular Biology,1994,5:25-40.
[17]东秀珠,蔡妙英.常见细菌系统鉴定手册.北京:科学出版社,2001:364-398.
[18]Margesin R. Permafrost soils. Berlin Heidelberg:Springer-Verlag,2009.
[19]Skidmore M,Anderson SP,Sharp M.Comparison of microbialcommunity compositions of two subglacial environments revealsa possible role formicrobesin chemical weathering processes.Applied and Environmental Microbiology,2005,71(11):6986-6997.
[20]Gangwar P,Alam SI,Bansod S,Singh L.Bacterial diversity of soil samples from the western Himalayas,India.Can J Microbiology,2009,55(5):564-577.
[21]Olivera NL,Sequeiros C,Nievas ML. Diversity and enzyme properties of protease-producing bacteria isolated from sub-Antarctic sediments of Isla de Los Estados,Argentina.Extremophiles,2007,11(3):517-526.
[22]Kim EH,Cho KH,Lee YM,Yim JH,Lee HK,Cho JC,Hong SG.Diversity ofcold-active protease-producing bacteria from arctic terrestrial and marine environments revealed by enrichment culture. The Journal of Microbiology,2010,48(4):426-432.
[23]Haruta S,Kato S,Yamamoto K,Igarashi Y.Intertwined interspeciesrelationships: approachestountanglethe microbial network.Environmental Microbiology,2009,11(12):2963-2969.
[24]Rossello-Mora R,Amann R.The species concept for prokaryotes.FEMS Microbiology Reviews,2001,25(1):39-67.
[25]Xu Y,Wang D,Shi X,Zheng X,Zhou H,Liu Ya,Ni Y.Selective isolation and diversity of cold-adapted lipaseproducing strains from permafrost soil at the terminus of a glacier in the Tianshan Mountains.Acta Microbiologica Sinica,2011,52(2):233-240.(in Chinese)徐宇丽,王大伟,史学伟,郑晓吉,周红,刘娅,倪永清.天山冻土产低温脂肪酶菌株的筛选及其多样性分析.微生物学报,2011,51(2):233-240.
[26]Zhang M,Gu Y,Xu Y,Shi X,Zheng X,Zhou H,Ni Y.Phylogenetic and physiologicaldiversity ofcoldadapted bacteria producing β-galactosidase from permafrost sediments of the bottom layer of the Glacier No.1 in the Tianshan Mountains.Acta Microbiologica Sinica,2011,51(12):1605-1615.(in Chinese)张明,顾燕玲,徐宇丽,史学伟,郑晓吉,周红,倪永清.天山1号冰川底部沉积层产β-半乳糖苷酶低温菌株的系统发育分析及生理多样性研究.微生物学报,2011,51(12):1605-1615.
[27]Susana CV, Silvia HC, WalterPMC. Extracellular proteases from eight psychrotolerant Antarctic strains.Microbiological Research,2004,159(2):157-166.
[28]Cho JC,Tiedje JM.Biogeography and degree of endemicity of fluorescent Pseudomonas strains in soil.Applied and Environmental Microbiology,2000,66(12):5448-5456.
[29]Rosselló-Mora R,Lucio M ,Peña A ,Brito-Echeverría J,López-López A ,Valens-Vadell M,Frommberger M,Antón J, Schmitt-Kopplin P. Metabolic evidence for biogeographic isolation ofthe extremophilic bacterium Salinibacter ruber.ISME Journal,2008,2(3):242-253.
Phylogenetic and physiological diversity of cold-adapted bacteria producing protease from sediments of the bottom layer of the Glacier No.1 in the Tianshan Mountains
Abstract:[Objective] We characterized proteolytic bacteria isolates from sediments of the bottom layer of the Glacier No.1 in the Tianshan Mountains,China.Physiological test and phylogenetic analysis were undertaken to expand our knowledge on diversity and ecological distribution of psycrotrophic and psycrophlic bacteria populations.[Methods] Using the screening media containing skim milk,we screened cold-adapted strains producing protease.Taxonomic identity and genetic variability of strains isolated was determined by partial 16S rRNA gene sequences and repetitive-element PCR fingerprint.[Results] Of the total 125 cold-adapted bacterial isolates,high levels of protease activity were observed from 27 isolates at optimal growth temperatures ranging from 15 to 24℃ in plate assay.Among 27 protease-producing strains,only 6 isolates were psychrophilic.The 16S rRNA gene phylogenetic analysis revealed that protease-producing isolates belonged to 5 phyum,namely α、,β and γof Proteobacteria,Actinobacteria and Cytophaga-Flexibacter-Bacteroides.They are affiliated tothe genera Pseudomonas, Polaromonas, Brevundimonas, Rholococces, Cryobacterium, Kocuria,Arthrobacter,Chryseobacterium and Flavobacterium.The populations of the predominant cultivated protease-producing bacteria are the Pseudomonas spp.(40.7%).[Conclusion] The results enriched our knowledge on the phylogenetic and physiological diversity of cold-adapted strains producing protease in cold environments.
Keywords:permafrost,Tianshan Mountains,bacterial phylogeny,cold-adapted protease
Supported by the National Natural Science Foundation of China(40961002,41140009),by the General and Special Program of the Postdoctoral Science Foundation of China(201003339)and by the Doctor Special Fund from the Xinjiang Production and Construction Corps(2011BB009)
*Corresponding author.Tel:+86-931-4967818;E-mail:gdcheng@lzb.ac.cn
Received:31 August 2012/Revised:9 November 2012
中图分类号:X172
文献标识码:A
文章编号:0001-6209(2013)02-0164-09
基金项目:国家自然科学基金项目(40961002;41140009);冻土工程国家重点实验室开放基金项目(SKLFSE200903);中国博士后科学基金特别资助(20100339);新疆生产建设兵团博士资金专项(2011BB009)
* 通信作者。E-mail:gdcheng@lzb.ac.cn
收稿日期:2012-08-31;
修回日期: 2012-11-09
