天山乌源1号冰川表面冰尘及底部沉积层真菌群落结构比较及其系统发育分析
【类型】期刊
【作者】王叙贤,顾燕玲,倪雪姣,关波,倪永清(石河子大学食品学院;中国科学院西北生态环境资源研究院冻土工程国家重点实验室)
【作者单位】石河子大学食品学院;中国科学院西北生态环境资源研究院冻土工程国家重点实验室
【刊名】冰川冻土
【关键词】 天山冰川;冰尘;沉积层;真菌;系统发育
【资助项】国家自然科学基金项目(41271268,40961002,41140009);冻土工程国家重点实验室开放基金项目(SKLFSE200903);中国博士后科学基金项目(20100339...
【ISSN号】1000-0240
【页码】P781-791
【年份】2019
【期号】第4期
【期刊卷】1;|7;|8;|2
【摘要】对天山乌源1号冰川表面冰尘(Fz)及底部沉积层(Dz)的理化性质及真菌的多样性、群落结构、OTU的系统发育进行了比较分析。ITS克隆文库分析显示,依据99%相似性共得到26个真菌OTU,表面冰尘和底部沉积层分别为21和22个,其中17个为共有OTU。天山1号冰川中真菌以担子菌门(Basidiomycota)、子囊菌门(Ascomycota)占绝对优势,子囊菌门的真菌在Fz、Dz文库中的比例分别约为27%、57%。系统发育分析显示子囊菌门的真菌有14个OUT,8个属,其中Cladosporium仅分布在冰川表面(6.25%),而Aureobasidium仅出现冰川底部沉积层(8.93%)。另外6个属Tetracladium、Pseudeurotium、Fusarium、Penicillium、Simplicillium、Aspergillus在冰川底部和表面均有分布,但丰度明显不同,尤其是Aspergillus、Simplicillium在冰川底部沉积物中占优势(分别为17.86%、12.50%)。担子菌门的克隆主要来自Rhodotorula、Leucosporidium属,前者主要在分布冰川表面(12%),底部沉积层很少,后者在冰川表面及底部均有分布,分别占文库的比例为8.69%、12.50%。冰川表面和底部沉积层生态条件迥异,对其中的微生物系统发育类群具有明显的选择性。
【全文】 文献传递
天山乌源1号冰川表面冰尘及底部沉积层真菌群落结构比较及其系统发育分析
摘 要: 对天山乌源1号冰川表面冰尘(Fz)及底部沉积层(Dz)的理化性质及真菌的多样性、 群落结构、 OTU的系统发育进行了比较分析。ITS克隆文库分析显示, 依据99%相似性共得到26个真菌OTU, 表面冰尘和底部沉积层分别为21和22个, 其中17个为共有OTU。天山1号冰川中真菌以担子菌门(Basidiomycota)、 子囊菌门(Ascomycota)占绝对优势, 子囊菌门的真菌在Fz、 Dz文库中的比例分别约为27%、 57%。系统发育分析显示子囊菌门的真菌有14个OUT, 8个属, 其中Cladosporium仅分布在冰川表面(6.25%), 而 Aureobasidium仅出现冰川底部沉积层(8.93%)。另外6个属Tetracladium、 Pseudeurotium、 Fusarium、 Penicillium、 Simplicillium、 Aspergillus在冰川底部和表面均有分布, 但丰度明显不同, 尤其是Aspergillus、 Simplicillium在冰川底部沉积物中占优势(分别为17.86%、 12.50%)。担子菌门的克隆主要来自Rhodotorula、 Leucosporidium属, 前者主要在分布冰川表面(12%), 底部沉积层很少, 后者在冰川表面及底部均有分布, 分别占文库的比例为8.69%、 12.50%。冰川表面和底部沉积层生态条件迥异, 对其中的微生物系统发育类群具有明显的选择性。
关键词: 天山冰川; 冰尘; 沉积层; 真菌; 系统发育
0 引言
真菌种类极其繁多,一般包括五大类群: 担子菌门、 子囊菌门、 接合菌门、 鞭毛菌门和壶菌门。最新研究统计, 全世界的真菌种类大约有150万, 但至今详细描述的却只有5%~10%[1], 绝大多数类群是未知的。一方面由于培养技术的局限性和不成熟, 缺乏适合于分离培养各种真菌种群的培养基和培养条件, 另一个原因是对真菌的栖息环境和代谢状况的了解并不全面[2]。真菌生态分布也极其广泛, 不仅栖息于农田、 森林、 草地、 沼泽湿地、 温泉热土中, 也频繁出现在动、 植物体表内外[3], 还能够适应各种极端环境而繁衍, 包括严寒的极地[4]、 高山冰川环境[5]、 冻土层[6]等。
由于不同地理差异和环境因子的影响, 在冰川不同的生态景观中微生物形成独特的群落结构、 组成及分布规律。冰川底部由冰、 水、 岩沉积层构成, 为缺氧、 缺光照、 高静压、 持续低温(0 ℃之下)极端严酷的生态系统, 是一个原始的、 较少受到外源环境影响的寡营养生态环境。研究表明冰川底部生境栖息着多样的原核细菌, 包括介导各种矿物质氧化还原过程的自养功能微生物类群和异养细菌[7], 在冰川新近退缩后植物定植之前, 这些微生物功能类群对冰碛石石和冰碛沉积物的风化、 冰川水化学、 土壤的前期发育起着关键作用[8-9]。与冰川底部环境相比, 冰川表面总体生态特征是: 气温昼夜和季节波动频繁、 紫外辐射较强、 氧气相对充足、 有机质丰富[10]。研究表明, 来自陆地的灰尘、 海洋表面气溶胶和火山灰烬等粘附在积雪、 矿物质或者有机质颗粒上沉降在冰川表面, 同时携带外源微生物进入冰川生态系统, 一些微生物代谢产生大量的胞外多糖, 通过粘附作用在冰川表面形成深色的堆状冰尘(cryoconite), 冰尘吸收太阳辐射能量后, 在冰川表面形态冰尘空洞, 积累丰富的有机质, 不仅是初级生产者光合微生物(藻类、 蓝细菌)栖息的场所, 也为各种异养细菌、 真菌、 原生生物(包括硅藻类、 纤毛虫类)的繁衍提供了生存环境[11]。其中, 真菌作为分解者通过分泌胞外酶, 能够降解木质纤维素等难降解碳源, 为藻类和异养细菌的生长繁殖提供易被吸收利用的养分, 因此真菌在维持冰川生态系统平衡方面发挥重要作用[12]。然而, 目前对冰川微生物的研究主要集中在细菌多样性、 分布格局及其系统发育, 影响细菌群落组成的环境因素, 以及冰川表面、 底部发生的生物地球化学过程[13-14], 而对于冰川环境中真菌群落结构及生态功能的报道较少。
天山乌源1号冰川是我国气象变化、 水文径流量、 物质平衡等数据最为完备的冰川[15-16]。目前有关天山1号冰川周边冻土、 积雪、 冰芯细菌、 冰尘蓝细菌多样性已有研究报道[14,17]。张威等[18]对天山1号冰川周边雪坑中的真核微生物多样性及时空变化进行了研究。对于冰川生态系统, 冰雪消融会形成自上而下的融水径流, 冰川表面冰尘、 底部沉积层及前沿等不同景观的微生物群落结构和生态学过程可能存在耦联。因此, 本文对天山1号冰川表面冰尘和底部沉积层中真菌群落的结构组成及系统发育类群差异进行了分析, 旨在解析生态环境条件截然不同, 但相互耦联的上、 下两个生态系统是否对微生物菌群具有选择作用, 同时为冰川环境中有潜在利用价值的耐低温真菌菌种资源的挖掘提供理论参考。
1 材料和方法
1.1 样品采集与处理
2014年9月5日, 于天山一号冰川西支表面, 海拔为3 833 m~4 480 m(43°06′ N, 86°49′ E)处, 设置3个取样点, 每个样点2 m×2 m, 取平行样5份; 在冰川末端消融区设置三个位点, 每个样点取样5份。将冰尘颗粒和去掉大块冰碛物的沉积物迅速装入己灭菌的保鲜盒内, 置于车载冰箱中-4~-8 ℃保存, 6 h之内运回实验室, 部分样品保存在-70 ℃超低温冰箱; 用于构建克隆文库的样品暂放-20 ℃保存, 取部分样品进行理化性质测定。以上所有过程均在无菌条件下完成。
1.2 培养基
用于构建克隆文库的培养基主要为SOB培养基(g·L-1) (酵母提取物5 g, 胰蛋白胨20 g, NaCl 0.58 g, 加10 mL 250 mmol·L-1 KCl溶液, 溶解于800 mL去离子水中, 定容至1 L, 121 ℃高压蒸汽灭菌20 min, 加入5 mL灭菌的2 mol·L-1 MgCl2溶液)。所用抗生素为氨苄青霉素钠盐(100 mg·mL-1), X-Gal及IPTG试剂浓度分别为20 mg·mL-1、 24 mg·mL-1。
1.3 样品理化性质测定
天山一号冰川表面冰尘和底部沉积层样品的pH值(水∶样品=2.5∶1)、 速效磷、 速效钾、 硝态氮、 总有机质及全氮、 全磷、 全钾的测定参照文献[19-20]进行, 每个样品测3次。
1.4 样品总DNA的提取及纯化
采用直接法结合商业试剂盒提取样品总DNA。直接法参照Zhou等[21]DNA提取法提取, 略有改动。5 g样品中加入DNA裂解液、 蛋白酶K及溶菌酶后, 在37 ℃、 220 r·min-1条件下水平震荡30 min; 65 ℃水浴2 h后加入20% SDS溶液, 超低温冰箱冻融三次后在水平击打器上击打10 min; 用苯酚、 氯仿、 异戊醇(25∶24∶1)离心抽提DNA, 异丙醇过夜沉淀DNA; 70%预冷酒精洗涤两次, 最后溶解于100 μL的TE溶液, 共提取3份样品, 用Wizard DNA Clean-Up System试剂盒纯化提取的总DNA。同时采用PowerSoilTM DNA Isolation Kit (Mo Bio Laboratories, Carls bad, CA, USA)试剂盒提取DNA, 每个样点提取5 份, 混合后用于文库构建。提取DNA保存在4 ℃条件下, 采用分光光度计对其核酸浓度和纯度进行定量。
1.5 样品真菌ITS基因间隔序列的PCR扩增
将3份冰川表面冰尘及底部沉积层样品总DNA分别混合后, 采用真菌ITS区特异引物ITS1F (5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS4 (5′-TCCTCCGCTTATTGATATGC-3′)扩增[22], 每个样品设5个平行样。扩增体系: Mixture 12.5 μL, 引物各0.8 μL, DNA模板1 μL, 加灭菌ddH2O补足25 μL。热循环条件: 94 ℃预变性5 min; 94 ℃变性1 min, 65 ℃复性30 s, 72 ℃延伸30 s (2个循环); 94 ℃变性1 min, 60 ℃复性30 s, 72 ℃延伸30 s; 94 ℃变性1 min, 55 ℃复性30 s, 72 ℃延伸30 s (2个循环); 94 ℃变性1 min , 52 ℃复性 30 s, 72 ℃延伸40 s (28个循环); 最后72 ℃延伸8 min。PCR扩增产物用1%的琼脂糖凝胶电泳检测其大小。
1.6 PCR产物纯化和基因文库的构建
将冰尘样品和沉积层样品扩增的PCR产物混合后, 用QIAquick PCR purification kit (Qiagen, Hilden, Germany)试剂盒纯化。纯化后的产物经电泳检测大小后与pEASY-T1 Cloning Vector (TransGen, Beijing, China)载体连接克隆。挑取白色单菌落, 采用载体引物M13进行PCR扩增鉴定, 筛选片段大小正确的阳性克隆子进行测序。
1.7 测序及数据处理
两个文库随机挑选的203个阳性克隆子, 采用载体引物, 由上海美吉完成测序。采用BioEdit 7.0.5软件 (http://www.mbio.ncsu.edu/BioEdit/bioedit.html)手动校正序列, 统一序列方向和长度, 保存为FASTA格式文件, 采用Ribosomal Database Project (RDP) II数据库的CHIMERA_CHECK program (seqmatch/seqmatch_intro.jsp)检测嵌合体序列, 在后续分析中予以剔除。
采用CLUSTAL X 1.83软件对齐序列, 输出文件采用phylip-3.68的DNADIST程序产生距离矩阵, 输入DOTUR-1.53软件在99%的相似度标准水平上[23], 采用furthest-neighbor method归类和确定OTU数(或Phylotypes), 并计算Shannon和Simpson多样性指数, 以及Nonparametric richness estimates (chaol指数), 并进行文库稀有度曲线分析。根据公式C=(1-n·N-1)·100%(N为文库总克隆数; n为文库中仅出现一次的OTU数量)计算克隆文库的Coverage。
1.8 系统发育树构建
将隶属于每个OTU的代表序列在NCBI的BLAST以及RDP′s Naïve Bayesian Classifier (confidence threshold, 80%)中进行最相近序列的分类匹配, 并提交到GenBank数据库中。每个OTU代表克隆序列与来自GenBank、 RDP II数据库中的亲缘关系较近的真菌ITS基因序列采用CLUSTALX 1.83软件联配, 采用邻接法(neighbor-joining method), 在MEGA v.5.1[24]软件中用Tamura-Nei model构建进化树, 进化树采用Bootstrap法评估, 重复次数为1 000。
2 结果与分析
2.1 理化性质
冰川表面、 冰尘表面及冰川沉积物采样点如图1所示。经分析表明, 天山乌源1号冰川表面粉尘颗粒粒径约为0.34~3.35 mm, 在冰川表面分布相对均一, 没形成明显的堆状丘, pH约为7.0, 冰川底部沉积层土壤pH约为7.75。冰尘样品(编号为Fz)中速效磷、 速效钾、 硝态氮、 有机质以及全氮含量均显著高于冰川底部沉积层土壤(编号为Dz)(表1)。其中1号冰川表面冰尘的硝态氮及全氮含量是底部沉积层土壤的8~10倍, 速效磷、 速效钾及有机质含量达到3~5倍。只有全磷、 全钾含量与冰川底部环境相近。

图1 冰川表面(a)、 冰尘表面(b)及冰川末端沉积物(c)取样点
Fig.1 Photographs showing the sampling sites on the glacier surface (a) , supraglacial cryoconite (b) and subglacial sediments (c)
表1 冰川表面冰尘和底部沉积物的理化特性比较
Table 1 Physicochemical properties of supraglacial cryoconite and subglacial sediments

算法水分含量∗/%pH(水∶土=2.5∶1)速效磷/(mg·kg-1)速效钾/(mg·kg-1)硝态氮/(mg·kg-1)有机质含量∗/%全氮∗/%全磷∗/%全钾∗/%表面冰尘平均45.567.0211.94195.6088.118.4000.3800.1102.770标准方差12.450.453.4656.6832.484.3400.0700.0350.540底部沉积物平均32.457.752.57144.508.901.3700.0300.0723.550标准方差10.800.230.3232.653.540.3600.0120.0140.080
注: *表示干重条件下的理化特性。
2.2 真菌的多样性及群落结构
2.2.1 真菌的多样性
从构建的表面冰尘真菌文库(Fz)和底部沉积层真菌文库(Dz)中, 各挑选了110个阳性克隆子进行测序, 排除嵌合体序列以及测序质量较差的序列后, Fz文库及Dz文库分别得到93条、 110条有效序列。将得到的序列联配构建距离矩阵, 用DOTUR-1.53软件在99%相似性标准下进行OTU的划分, Fz、 Dz分别得到21、 22个OUT (GenBank KX214352-KX214394)。多样性指数统计结果显示Fz样品的Shannon、 Simpson指数分别为2.873、 0.133, 而Dz样品的Shannon、 Simpson指数分别为2.108、 0.152, 冰尘样品中真菌的多样性稍高于冰川底部沉积层(表2)。
表2 冰川表面冰尘及底部沉积层真菌文库多样性指数
Table 2 The diversity and richness estimates of the fungal ITS gene clone libraries from supraglacial cryoconite and subglacial sediments
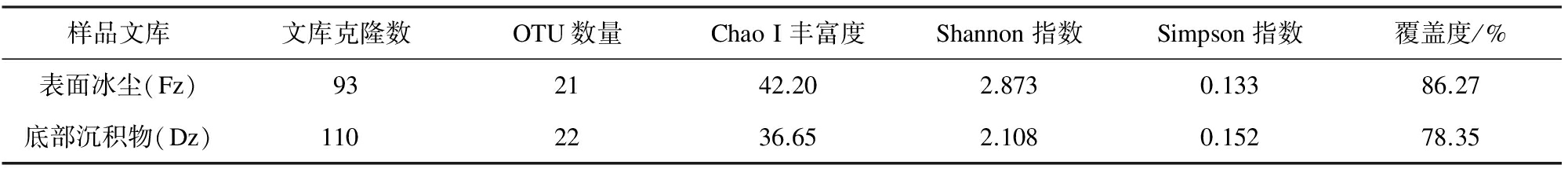
样品文库文库克隆数OTU数量ChaoI丰富度Shannon指数Simpson指数覆盖度/%表面冰尘(Fz)932142.202.8730.13386.27底部沉积物(Dz)1102236.652.1080.15278.35
依据OTU数和测序的有效克隆子数绘制稀有度曲线(图2), 当克隆子数大于100时, 两文库的曲线都已渐渐趋于平缓, 其盖度(coverage)分别达到了86.27%、 78.35%, 表明构建文库测序数目基本趋于饱和, 两个文库挑选的克隆子基本能够涵盖天山1号冰川表面冰尘及底部沉积层中绝大部分真菌群落, 也能够代表其真菌多样性。
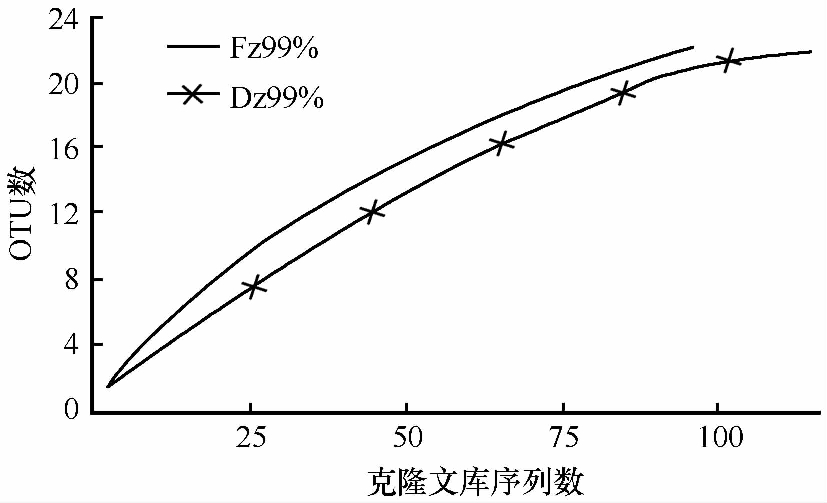
图2 冰川表面冰尘及底部沉积层真菌克隆文库稀疏曲线
Fig.2 Rarefaction curves constructed for fungal clone libraries from supraglacial cryoconite and subglacial sediments
2.2.2 真菌群落结构在门(phylum)水平上的差异
将Fz、 Dz文库得到的203条有效序列, 用DOTUR-1.53软件在99%相似性标准下进行OTU的划分, 两个文库所有序列被划分为26个OTU。利用BLAST、 RDP Classifier在线工具, 对每个OTU的代表序列进行最相近序列匹配, 根据匹配结果, 在门水平上分别统计Fz、 Dz两个文库的群落结构(图3)。统计显示, Fz真菌在门水平上隶属于三个系统发育类群: 担子菌门(Basidiomycota, 21.74%)、 子囊菌门(Ascomycota, 27.17%)、 壶菌门(Chytridiomycota, 2.17%)[图3(a)], 其余序列属于不能明确划分隶属关系的真菌(unclassified fungi), 占总文库克隆数的48.92%。Dz真菌也包括同样的系统发育类群: 担子菌门(Basidiomycota, 17.86%)、 子囊菌门(Ascomycota, 57.14%)、 壶菌门(Chytridiomycota, 7.14%)[图3(b)], 但只有18%的克隆序列属于不能明确划分隶属关系的真菌(unclassified fungus)。显然隶属于担子菌门和子囊菌门的序列在两个文库中均占绝对优势, 其中隶属于担子菌门的真菌在Fz、 Dz文库中的比例接近, 而属于子囊菌门的真菌在Fz、 Dz文库中的比例分别约为27%、 57%, 差异明显。
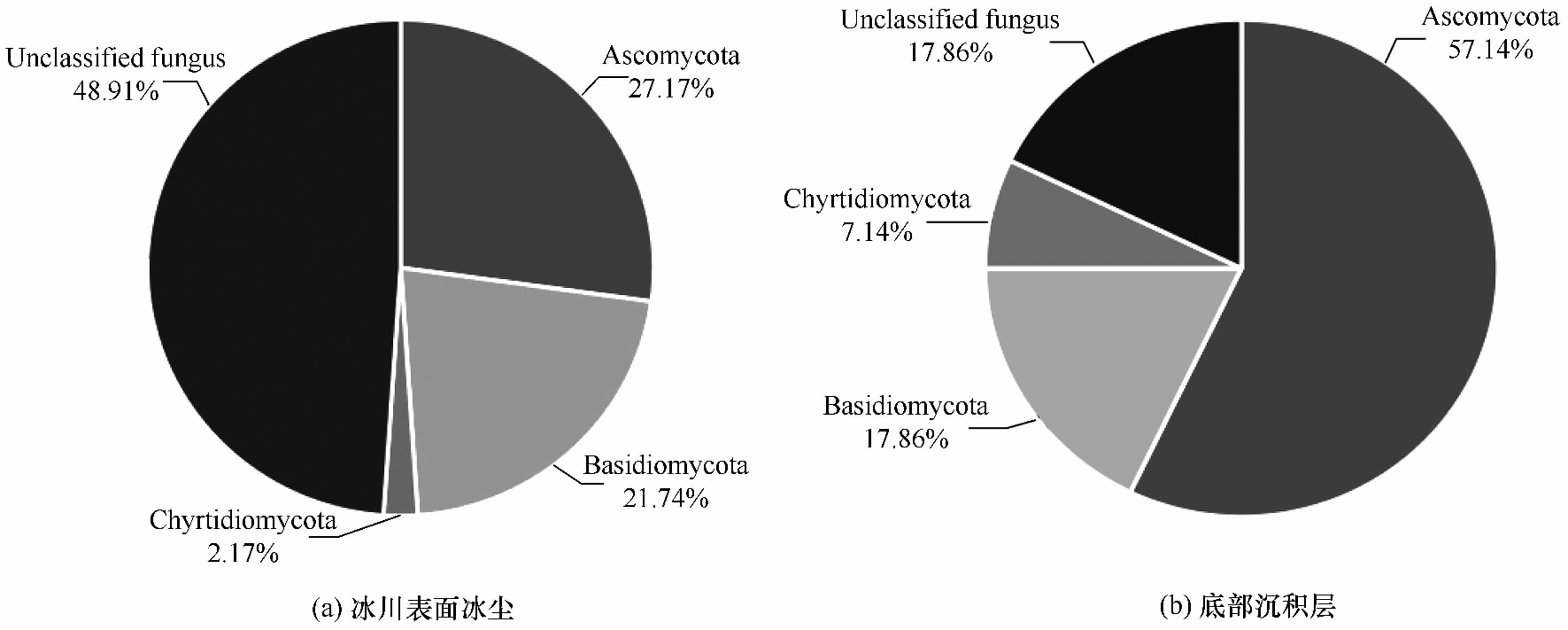
图3 真菌ITS区基因门水平群落结构组成
Fig.3 Relative abundance of the fungal phyla of ITS clone libraries from supraglacial cryoconite (a) and subglacial sediments (b)
2.2.3 真菌群落结构在属(genus)水平上的差异
对结果进一步统计显示, 一号冰川Fz、 Dz真菌ITS基因文库序列代表的真菌在属水平上的群落结构如图4。担子菌门的红酵母属(Rhodotorula)主要在分布冰川表面, 约占12%, 白冬孢酵母属(Leucosporidium)在冰川表面及底部均有分布, 分别为8.69%、 12.50%。
子囊菌门的真菌有9个属, 其中枝孢菌属(Cladosporium)仅分布在冰川表面(6.25%), 而金担子菌属(Aureobasidium)仅出现冰川底部沉积层(8.93%)。散囊菌属(Pseudeurotium)、 镰刀菌属(Fusarium)、 青霉菌属(Penicillium)、 且拟青霉属(Simplicillium) 曲霉属(Aspergillus)、 Tetracladium及一类不能准确鉴定的Ascomycota真菌等七个属在冰川底部和表面均有分布, 但丰度明显不同, 尤其是曲霉属(Aspergillus) 、 拟青霉属(Simplicillium)在冰川底部沉积物中占优势(分别为17.86%、 12.50%), 而在表面粉尘相对较少(分别为6.25%、 1.09%), 其它几个属的丰度相差较小。除此之外, 冰川表面不能明确划分隶属关系的OTU1和OTU7代表的真菌占群落总数的45%左右, 而冰川底部沉积物不能明确划分隶属关系的OTU1和OTU7在冰川底部只有3.64%, OTU19代表一个分类地位不能确定的真菌, 占比仅仅为1.09%, 显著低于冰川表面。属于壶菌门(Chytridiomycota)的只有一个属Rhizophydium, 在冰川底部沉积物中占7.14%, 表面粉尘只有2.17%。
2.3 真菌OTU系统发育及其最相近生态位
天山1号冰川表面冰尘(Fz)及底部沉积层(Dz)真菌ITS基因文库依据99%的相似性划分为26个OTU, 其中共有OTU 17个, Fz、 Dz特有OTU分别为4、 5个, 用每个OTU的代表序列以及数据库的最相近序列构建系统发育树(表3、 图5)。此外, 每个OTU的代表克隆序列与BLAST、 RDP数据库中最相近序列大多数来源于冰川等低温环境(表3)。
2.3.1 子囊菌门(Ascomycota)
26个OTU中, 属于子囊菌门的有14个, 隶属于9个属。OTU4、 OTU5、 OTU11、 OTU14的代表序列在系统发育上分别于Aspergillus oryzae、 Aspergillus versicolor、 Aspergillus flavus、 Aspergillus pseudoglaucus相似性达到99~100%, 其中OTU 14仅在冰川沉积层检测到。OTU3与子囊菌门的菌株相似性达到97%。OTU10、 OTU26的代表序列分别与来自青藏高原的菌株Tetracladium(KC180672)和耐冷的Tetracladium psychrophilum的相似性最高, 分别为99%和96%, 其中OTU26仅出现在冰川表面。以OTU8为代表的克隆子在Dz与Fz文库中均有分布, 与Pseudeurotium bakeri相似度达到了99%。OTU25的克隆子序列与植物叶表面的真菌Cladosporium sp.、 洞穴气生真菌Cladosporium cladosporioides的相似度达到99%~100%, 仅仅出现在冰川表面粉尘, 很可能来自大气粉尘中的花粉或者植物残渣。以OTU13为代表的10个克隆全部来自冰川底部Dz文库, 系统发育上与类酵母Aureo-basidium pullulans亲缘关系很近, 相似性为96%。OTU20、 OTU22代表的真菌分别只有5、 4个克隆子, 分别与印度某地土壤中分出的菌株Penicillium sp. P-1以及Fusarium sp. BAB-3397相似度达到99%。依据ITS序列99%的相似性cutoff值, 同一个属Simplicillium的克隆子被划分为两个OTU (OTU15和OTU24), 其中OTU15的7个克隆均来自冰川底部沉积物。
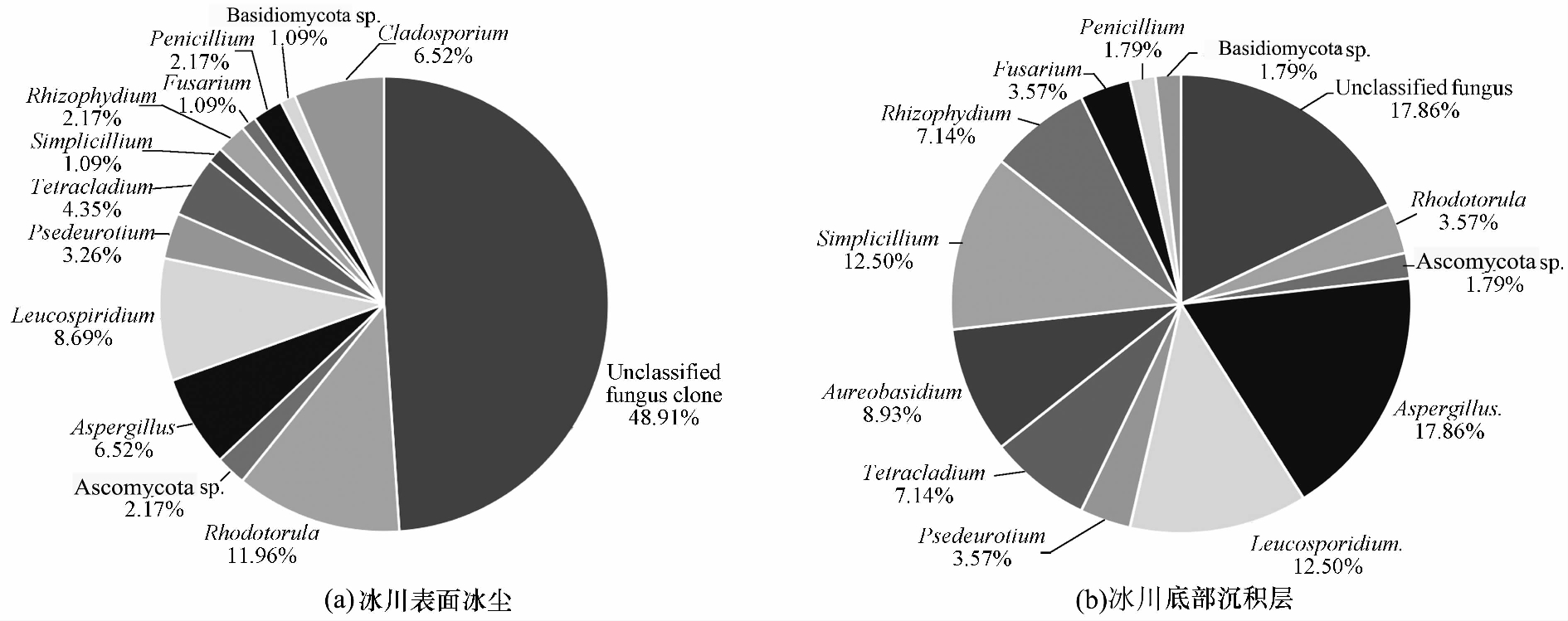
图4 真菌ITS基因文库中属水平上群落结构组成
Fig.4 Relative abundance of the fungal genus of ITS clone library from supraglacial cryoconite (a) and subglacial sediments (b)
表3 冰川表面冰尘及底部沉积层真菌克隆文库OTU的系统发育及最匹配地理来源
Table 3 The phylogenetic analysis and best BLAST match of OTUs of the fungal ITS clone libraries of supraglacial cryoconite and subglacial sediments
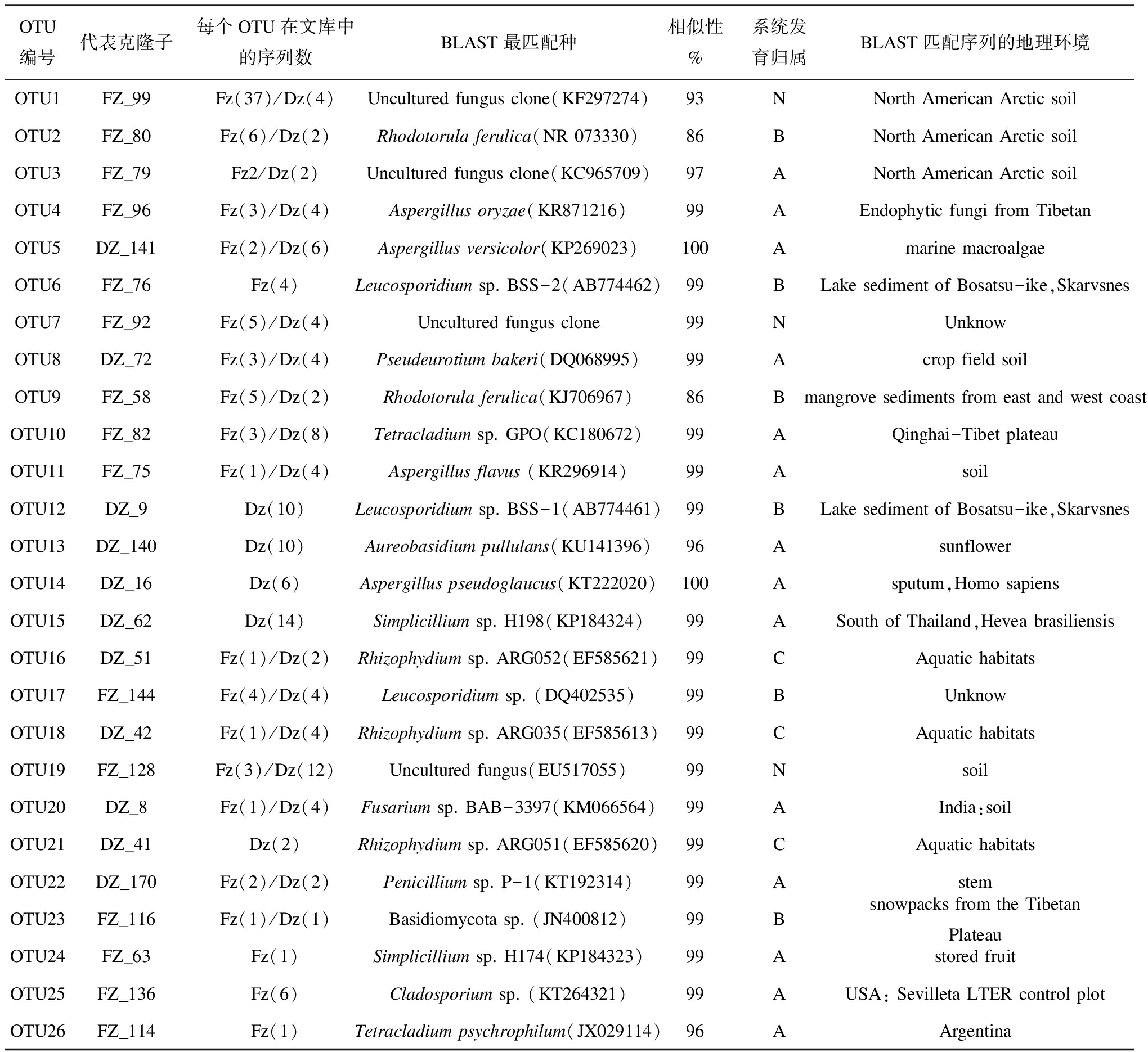
OTU编号代表克隆子每个OTU在文库中的序列数BLAST最匹配种相似性%系统发育归属BLAST匹配序列的地理环境OTU1FZ_99Fz(37)/Dz(4)Unculturedfungusclone(KF297274)93NNorthAmericanArcticsoilOTU2FZ_80Fz(6)/Dz(2)Rhodotorulaferulica(NR073330)86BNorthAmericanArcticsoilOTU3FZ_79Fz2/Dz(2)Unculturedfungusclone(KC965709)97ANorthAmericanArcticsoilOTU4FZ_96Fz(3)/Dz(4)Aspergillusoryzae(KR871216)99AEndophyticfungifromTibetanOTU5DZ_141Fz(2)/Dz(6)Aspergillusversicolor(KP269023)100AmarinemacroalgaeOTU6FZ_76Fz(4)Leucosporidiumsp.BSS-2(AB774462)99BLakesedimentofBosatsu-ike,SkarvsnesOTU7FZ_92Fz(5)/Dz(4)Unculturedfungusclone99NUnknowOTU8DZ_72Fz(3)/Dz(4)Pseudeurotiumbakeri(DQ068995)99AcropfieldsoilOTU9FZ_58Fz(5)/Dz(2)Rhodotorulaferulica(KJ706967)86BmangrovesedimentsfromeastandwestcoastOTU10FZ_82Fz(3)/Dz(8)Tetracladiumsp.GPO(KC180672)99AQinghai-TibetplateauOTU11FZ_75Fz(1)/Dz(4)Aspergillusflavus(KR296914)99AsoilOTU12DZ_9Dz(10)Leucosporidiumsp.BSS-1(AB774461)99BLakesedimentofBosatsu-ike,SkarvsnesOTU13DZ_140Dz(10)Aureobasidiumpullulans(KU141396)96AsunflowerOTU14DZ_16Dz(6)Aspergilluspseudoglaucus(KT222020)100Asputum,HomosapiensOTU15DZ_62Dz(14)Simplicilliumsp.H198(KP184324)99ASouthofThailand,HeveabrasiliensisOTU16DZ_51Fz(1)/Dz(2)Rhizophydiumsp.ARG052(EF585621)99CAquatichabitatsOTU17FZ_144Fz(4)/Dz(4)Leucosporidiumsp.(DQ402535)99BUnknowOTU18DZ_42Fz(1)/Dz(4)Rhizophydiumsp.ARG035(EF585613)99CAquatichabitatsOTU19FZ_128Fz(3)/Dz(12)Unculturedfungus(EU517055)99NsoilOTU20DZ_8Fz(1)/Dz(4)Fusariumsp.BAB-3397(KM066564)99AIndia:soilOTU21DZ_41Dz(2)Rhizophydiumsp.ARG051(EF585620)99CAquatichabitatsOTU22DZ_170Fz(2)/Dz(2)Penicilliumsp.P-1(KT192314)99AstemOTU23FZ_116Fz(1)/Dz(1)Basidiomycotasp.(JN400812)99BsnowpacksfromtheTibetanPlateauOTU24FZ_63Fz(1)Simplicilliumsp.H174(KP184323)99AstoredfruitOTU25FZ_136Fz(6)Cladosporiumsp.(KT264321)99AUSA:SevilletaLTERcontrolplotOTU26FZ_114Fz(1)Tetracladiumpsychrophilum(JX029114)96AArgentina
注: 系统发育归属中A为子囊菌门, B为担子菌门, C为壶菌门, N为不可鉴定。
2.3.2 担子菌门(Basidiomycota)
担子菌门的真菌在天山一号冰川表面冰尘及底部沉积层中属于第二大类, 包括6个OTU。OTU6、 OTU12、 OTU17为代表的22个克隆子分别与来自湖泊沉积层、 土壤的3个Leucosporidium sp.菌株的ITS序列相似度达到了99%(图3), 其中OTU6仅出现在表面粉尘, OTU12在冰川底部沉积物检测到, OTU17在冰川表面和底部均存在。OTU2和OTU9在系统发育上与酵母Rhodotorula ferulica亲缘关系较近, 但相似性只有86%, 而且两个OTU在冰川表面居多, 在冰川底部都仅仅检测到2个克隆子, 很可能代表新的分类单位。此外, 在BLAST匹配结果显示, OUT23的代表序列与来自青藏高原积雪中的一个菌株TP-Snow-Y1(JN400812)的ITS相似性达到99%, 但显然这个菌株并未鉴定到属, 很可能也代表一个新的分类单元。
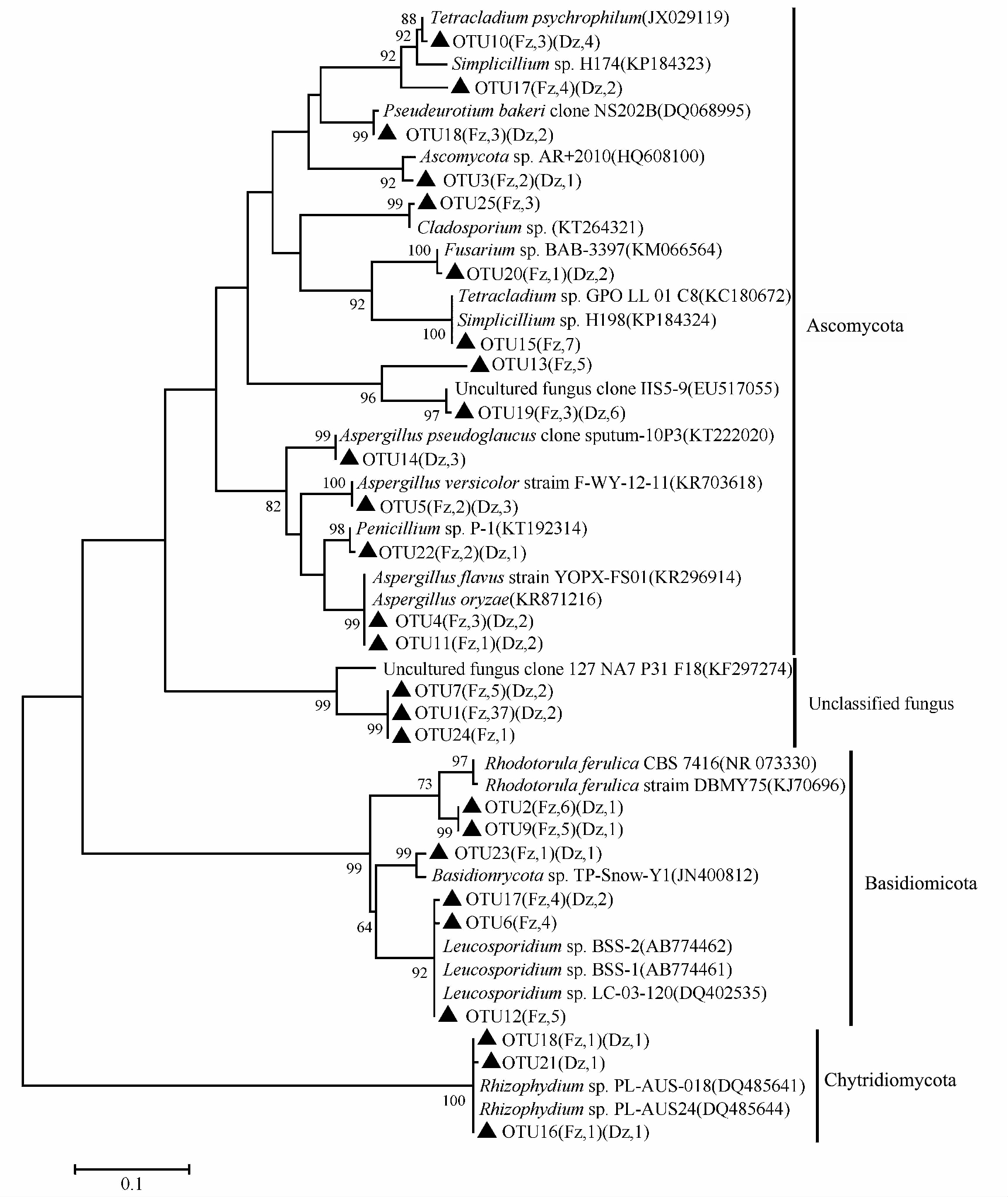
图5 Neighbor-joining法构建的天山乌源1号冰川真菌ITS基因文库序列的系统发育树
Fig.5 Neighbo-joining tree showing the phylogenetic relationships of OTUs of the fungal ITS clone libraries in supraglacial cryoconite and subglacial sediments of the Glacier No.1 in the headwaters of the Ürümqi River in the Tianshan Mountains and their closely related sequences downloaded from GenBank
2.3.3 壶菌门(Chytridiomycota)
天山一号冰川表面冰尘及底部沉积层中属于壶菌门的真菌极少, 检测结果显示代表序列均属于Rhizophydium, 依据99%的cutoff值, 分属于三个OTU, 分别为OTU16、 OTU18、 OTU21, 分别与来自阿根廷水生环境的三个序列相似度达到99%, 系统发育上隶属于Rhizophydium。
3 讨论
地球上大约11%陆地面积被冰川覆盖, 处于0 ℃ 以下的永久低温环境。来自冰川周边陆地的灰尘、 海洋表面气溶胶和火山灰烬等各种养分和微生物共同沉降在冰川表面, 微生物代谢产生大量的胞外多糖, 通过粘附作用在冰川表面形成深色的堆状冰尘, 冰尘吸收太阳辐射融进冰川表面形成冰尘空洞。研究表明, 冰尘和空洞有机质含量丰富, 栖息着作为初级生产者的光合微生物(绿藻类、 蓝细菌)[25]。最近有研究认为, 冰川表面进行光合自养过程的微生物仅仅贡献了小部分的有机碳、 氮养分, 其大部分养分来自大气尘埃、 颗粒的沉降积累作用[26]。本研究检测显示, 天山1号冰川表面冰尘样品中速效磷、 速效钾、 硝态氮、 有机质以及总氮含量均显著高于冰川底部沉积层, 其中硝态氮及总氮含量是底部沉积层土壤的8~10倍, 有机质含量达到4~5倍。众所周知, 真菌在自然界的物种多样性极其丰富, 具有极强的生态适应能力。作为分解者的真菌, 其最重要的生态学作用是通过产生菌丝体和分泌胞外酶降解环境中大部分细菌不能利用的木质纤维素等复杂的碳源[27]。因此, 在寡营养环境的冰川生态体系中, 真菌能够降解由大气尘埃、 火山灰烬沉降而累积的复杂有机质碳源, 对冰川物质循环、 生态平衡的维持发挥重要作用, 尤其为生物量较高的藻类和异养细菌的生长繁殖提供了易吸收利用的养分[28]。
冰川生态系统总体上是一个开放的体系, 因为在冰川表面积雪和冰层融水的冲刷作用下, 融水会携带积累的部分养分和微生物通过冰川表面的锅穴、 裂隙到达冰川底部, 为底部沉积层提供有机小分子, 作为有氧酵解或厌氧硫酸盐、 硝酸盐氧化还原作用的底物[29], 而冰川末端融水径流又会导出部分养分和微生物到冰川前沿生境, 因此冰川表面冰尘、 底部沉积层及前沿等不同景观的微生物生态学过程应该存在耦联, 群落结构存在一定程度的相似性。天山1号冰川中除了不能准确鉴定的OTU1、 OTU7、 OTU19代表的真菌外, 相对优势的属还包括丝状真菌Aspergillus、 Penicillium、 Cladosporium、 Tetracladium以及酵母菌Rhodotorula、 Leucosporidium, 此外也发现了类酵母属Aureobasidium的存在, 这些真菌类群在1号冰川表面和底部沉积层均存在。这个结果与格陵兰岛冰川表面冰尘中存在的丝状真菌类似, 包括子囊菌门的Penicillium、 Aspergillus、 Cephalothecium、 Circinella、 Alternaria[30]; 此外, 依据培养分离法, 对于欧洲、 亚洲、 南北两极的冰川表面及底部沉积物的酵母菌进行了广泛的研究报到, 最常见的酵母菌包括Cryptococcus、 Rhodotorula、 Mrakia, 以及隶属于子囊菌门的类酵母属Aureobasidium[31]。本研究同样检测到Rhodotorula、 Aureobasidium, 同时笔者采用培养法发现1号冰川表面、 底部、 融水得到的培养物中Cryptococcus也是最常见的类群(另撰文报道), 但是奇怪的是本文并没有检测到Cryptococcus。其原因一是在最广泛使用的培养基上Cryptococcus可能是最容易分离的酵母菌类群, 但其丰度在冰川生境中实际并不高; 另外一个原因是本研究采用的文库克隆法测序通量较小, 目前笔者正在采用高通量测序对冰川不同生境及其前沿环境的真菌多样性进行详细分析。
最近, 越来越多的证据显示, 无论在群落(community)水平上还是群体(population)水平上, 微生物的分布均受到生态环境因素的显著影响, 即微生物同高等动植物一样, 同样占据特定的生态位, 特殊环境条件的选择驱动微生物分类单位构成不同的群落结构[32]。冰川表面生态条件和底部沉积层存在巨大反差, 冰川底部缺氧、 缺光照、 高静压、 持续低温(0 ℃之下), 而冰川表面虽然也是相对低温, 但昼夜、 季节温度频繁波动、 紫外辐射较强、 氧气相对充足、 有机质丰富[25]。研究显示, 冰川生态系统相对苛刻的环境条件对生存繁衍于其中的活性功能微生物类群具有明显的选择性。我们的前期研究显示, 天山1号冰川表面粉尘中蓝细菌文库序列代表的属Leptolyngbya、 Oscillatoria和Phormidium同样在南北极冰冻圈环境中广泛存在, 与南北极得到的培养物及克隆序列亲缘关系最近, 意味着冰川表面对微生物的选择作用, 具有很强的温度和湿度剧烈波动耐受性、 抗紫外、 适合寡营养的种属才能在冰川表面定植, 而在冰川底部没有检测到这些蓝细菌的存在[17]。本研究发现, 隶属于同一个真菌属不同的OTU, 有可能出现在冰川表面或者底部不同生境, 譬如OTU6、 OTU12、 OTU17为代表的22个克隆子均属于Leucosporidium, 但OTU6仅出现在表面粉尘, OTU12仅仅在冰川底部沉积物中能被检测到, OTU17在冰川表面和底部均存在; OTU2和OTU9属于Rhodotorula, 主要存在于冰川表面; OTU25的6克隆子序列代表Cladosporium, 全部来自冰川表面粉尘; 以OTU13为代表的10个克隆与Aureobasidium pullulans亲缘关系很近, 全部来自冰川底部, OTU15的14克隆也全部来自冰川底部。除此之外, 冰川表面不能明确划分隶属关系的OTU1和OTU7代表的真菌(45%左右)在冰川底部沉积物丰度很低(只有3.64%)。因此, 冰川不同生态系统很可能对微生物近缘种群也具有选择性。然而, 目前基于rRNA基因免培养的多样性研究方法本身的缺陷以及缺乏对这些种群的广泛分离培养, 目前仍然不能肯定这些丝状真菌和酵母菌种群的地理分布是否具有冰冻圈生境特异性(具有特殊的基因型、 生态型及相对应的表型), 尤其是一些亲缘关系很近的、 功能类似的群体内部, 在冰川生态条件截然不同的小尺度环境中是否类似于海洋浮游蓝细菌Prochlorococcus spp.种群——对光强度、 温度等环境梯度的适应性而呈现不同的生态位分布格局[33], 这些问题都有待进一步深入研究。
此外, 天山1号冰川上空的大气环流、 降雨量、 降雪覆盖的时间、 风向都会影响表面粉尘颗粒的数量及物理化学条件。已经有研究表明, 乌源冰川表面粉尘在5、 6月由于沙尘活动颗粒浓度升高, 7、 8月由于季节性大气环境的变化和降水的作用, 表面粉尘被洗刷, 颗粒的矿物含量会发生变化[34], 因此有必要针对季节变化取样, 进一步阐明真菌群落结构的时间演替规律, 对阐明冰川生态系统的生物地球化学循环具有重要意义。
4 结论
天山乌源1号冰川表面冰尘(Fz)及底部沉积层(Dz)真菌以担子菌门(Basidiomycota)、 子囊菌门(Ascomycota)占绝对优势, 不能归类的真菌所占比很高。共得到26个真菌OTU, 其中17个为共有OTU。系统发育分析显示, 子囊菌门真菌8个属, 其中6个属在冰川底部和表面均有分布。冰川表面和底部沉积层生态条件迥异, 对其中的真菌系统发育类群具有明显的选择性。
参考文献(References):
[1] Taylor D L, Hollingsworth T N, McFarland J, et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning[J]. Ecological Monographs, 2014, 84(1): 3-20.
[2] Hawksworth D L. Global species numbers of fungi: are tropical studies and molecular approaches contributing to a more robust estimate?[J]. Biodiversity Conservation, 2012, 21(9): 2425-2433.
[3] Hawksworth D L. The magnitude of fungal diversity: the 1.5 million species estimate revisited[J]. Mycological Research, 2001, 105(12): 1422-1432.
[4] Arenz B E, Blanchette R A. Distribution and abundance of soil fungi in Antarctica at sites on the Peninsula, Ross Sea Region and McMurdo Dry Valleys[J]. Soil Biology and Biochemistry, 2011, 43(2): 308-315.
[5] Jumpponen A. Soil fungal community assembly in a primary successional glacier forefront ecosystem as inferred from rDNA sequence analyses[J]. New Phytologist, 2003, 158(3): 569-578.
[6] Schadt C W, Martin A P, Lipson D A, et al. Seasonal dynamics of previously unknown fungal lineages in tundra soils[J]. Science, 2003, 301(5638): 1359-1361.
[7] Skidmore M L, Foght J M, Sharp M J, et al. Microbial life beneath a high arctic glacier[J]. Applied and Environmental Microbiology, 2000, 66(8): 3214-3220.
[8] Sharp M, Parkes J, Cragg B, et al. Widespread bacterial populations at glacier beds and their relationship to rock weathering and carbon cycling[J]. Geology, 1999, 27(2): 107-110.
[9] Skidmore M, Anderson S P, Sharp M, et al. Comparison of microbial community compositions of two subglacial environments reveals a possible role for microbes in chemical weathering processes[J]. Applied and Environmental Microbiology, 2005, 71(11): 6986-6997.
[10] Edwards A, Anesio A M, Rassner S M, et al. Possible interactions between bacterial diversity, microbial activity and supraglacial hydrology of cryoconite holes in Svalbard[J]. The ISME Journal, 2011, 5(1): 150-160.
[11] Vincent W F. Evolutionary origins of antarctic microbiota: invasion, selection and endemism[J]. Antarctic Science, 2000, 12(3): 374-385.
[12] Takeuchi N, Matsuda Y, Sakai A, et al. A large amount of biogenic surface dust(cryoconite) on a glacier in the Qilian Mountains, China[J]. Bulletin of Glaciological Research, 2005, 22(22): 1-8.
[13] Li Mingyuan, Wang Jilian, Gulbahar Sawut. Culturable bacterial diversity in snow, ice and meltwater of the Yangbark Glacier, Muztag Ata[J]. Journal of Glaciology and Geocryology, 2015, 37(6): 1634-1641. [李明源, 王继莲, 古丽巴哈尔·萨吾提. 新疆东帕米尔高原慕士塔格峰洋布拉克冰川雪冰及融水中可培养细菌多样性分析[J]. 冰川冻土, 2015, 37(6): 1634-1641.]
[14] Tao Ling, Gu Yanling, Zheng Xiaoji, et al. Phylogeny and physiological diversity of bacteria from meltwater of the Glacier No.1 in the Tianshan Mountains[J]. Journal of Glaciology and Geocryology, 2015, 37(2): 511-521. [陶玲, 顾燕玲, 郑晓吉, 等. 天山一号冰川融水可培养细菌生理生化特性及其系统发育[J]. 冰川冻土, 2015, 37(2): 511-521.]
[15] Sun Meiping, Li Zhongqin, Yao Xiaojun, et al. Analysis on runoff variation of Glacier No.1 at the headwaters of the Ürümqi River from 1959 to 2008[J]. Journal of Natural Resources, 2012, 27(4): 650-660. [孙美平, 李忠勤, 姚晓军, 等. 1959-2008年乌鲁木齐河源1号冰川融水径流变化及其原因[J]. 自然资源学报, 2012, 27(4): 650-660.]
[16] Li Zhongqin, Shen Yongping, Wang Feiteng, et al. Response of glacier melting to climate change: take Ürümqi Glacier No.1 as an example[J]. Journal of Glaciology and Geocryology, 2007, 29(3): 333-342. [李忠勤, 沈永平, 王飞腾, 等. 冰川消融对气候的影响: 以乌鲁木齐河源1号冰川为例[J]. 冰川冻土, 2007, 29(3): 333-342.]
[17] Ni Xuejiao, Qi Xing′e, Gu Yanling, et al. Community structure and phylogenetic analysis of cyanobacteria in cryoconite from surface of the Glacier No.1 in the Tianshan Mountains[J]. Acta Microbiologica Sinica, 2014, 54(11): 1256-1266. [倪雪姣, 齐兴娥, 顾燕玲, 等. 天山乌鲁木齐河源一号冰川表面粉尘蓝细菌群落结构及其系统发育[J]. 微生物学报, 2014, 54(11): 1256-1266.]
[18] Zhang Wei, Zhang Gaosen, Liu Guangxiu, et al. Diversity and its temporal-spatial characteristics of eukaryotic microorganisms on Glacier No.1 at the Ürümqi river head, Tianshan Mountains[J]. Journal of Glaciology and Geocryology, 2010, 32(5): 906-913. [张威, 章高森, 刘光琇, 等. 天山乌鲁木齐河源1号冰川中真核微生物多样性分布及时空变化研究[J]. 冰川冻土, 2010, 32(5): 906-913.]
[19] Stibal M, Wadham J L, Lis G P, et al. Methanogenic potential of Arctic and Antarctic subglacial environments with contrasting organic carbon sources[J]. Global Change Biology, 2012, 18(11): 3332-3345.
[20] Boyd E S, Lange R K, Mitchell A C, et al. Diversity, abundance, and potential activity of nitrifying and nitrate-reducing microbial assemblages in a subglacial ecosystem[J]. Applied and Environmental Microbiology, 2011, 77(14): 4778-4787.
[21] Zhou Jizhong, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology, 1996, 62(2): 316-322.
[22] Anderson I C, Cairney J W. Diversity and ecology of soil fungal communities: increased understanding through the application of molecular techniques[J]. Environmental Microbiology, 2004, 6(8): 769-779.
[23] Schloss P D, Handelsman J. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness[J]. Applied and Environmental Microbiology, 2005, 71(3): 1501-1506.
[24] Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[25] Hodson A, Anesio A M, Tranter M, et al. Glacial ecosystems[J]. Ecological Monographs, 2008, 78(1): 41-67.
[26] Stibal M, Tranter M, Benning L G, et al. Microbial primary production on an Arctic glacier is insignificant in comparison with allochthonous organic carbon input[J]. Environmental Microbiology, 2008, 10(8): 2172-2178.
[27] Treseder K K, Lennon J T. Fungal traits that drive ecosystem dynamics on land[J]. Microbiology and Molecular Biology Reviews, 2015, 79(2): 243-262.
[28] Anderson S P. Biogeochemistry of glacial landscape systems[J]. Annual Review of Earth and Planetary Sciences, 2007, 35(1): 375-399.
[29] Stibal M,  abacká M, Žársk
abacká M, Žársk J. Biological processes on glacier and ice sheet surfaces[J]. Nature Geoscience, 2012, 5(11): 771-774.
J. Biological processes on glacier and ice sheet surfaces[J]. Nature Geoscience, 2012, 5(11): 771-774.
[30] Christner B C, Kvitko B H, Reeve J N. Molecular identification of bacteria and eukarya inhabiting an Antarctic cryoconite hole[J]. Extremophiles, 2003, 7(3): 177-183.
[31] Buzzini P, Branda E, Goretti M, et al. Psychrophilic yeasts from worldwide glacial habitats: diversity, adaptation strategies and biotechnological potential[J]. FEMS Microbiology Ecology, 2012, 82(2): 217-241.
[32] Hanson C A, Fuhrman J A, Horner-Devine M C, et al. Beyond biogeographic patterns: processes shaping the microbial landscape[J]. Nature Review Microbiology, 2012, 10(7): 497-506.
[33] Johnson Z I, Zinser E R, Coe A, et al. Niche partitioning among prochlorococcus ecotypes along ocean-scale environmental gradients[J]. Science, 2006, 311(5768): 1737-1740.
[34] Dong Zhiwen, Li Zhongqin, Wang Feiteng, et al. Characteristics of atmospheric dust deposited in snow on Ürümqi Glacier No.1 of eastern Tian Shan, China: a comparison of measurements during Asian dust period with non-dust period[J]. Environmental Science, 2009, 30(6): 1818-1825. [董志文, 李忠勤, 王飞腾, 等. 天山乌鲁木齐河源冰川积雪内不溶粉尘特征: 沙尘与非沙尘活动季节的比较[J]. 环境科学, 2009, 30(6): 1818-1825.]
(本文编辑: 庞瑜)
Composition and phylogeny of fungal community in supraglacial cryoconite and subglacial sediments of the Glacier No.1 at headwaters of the Ürümqi River in Tianshan Mountains
Abstract: The diversity, composition and phylogeny of fungal community in supraglacial cryoconites and subglacial sediments of the Glacier No.1 in the headwaters of the Tianshan Mountains, China are assessed. Analysis of amplified rDNA ITS region sequences allows grouping of 203 clones into 26 operational taxonomic units (OTUs) using a cut-off of 99% identity. Twenty-two and twenty-one OTUs were found in the subglacial and supraglacial clone libraries, respectively. BLAST searches show that the subglacial community of the glacier was dominated by representatives of phylum Ascomycota (57% of clones), followed by phylum Basidiomycota (18% of clones) and Chytridiomycota (7% of clones), with the remainder of the library belonging to unclassified fungal OTUs (18% of clones). The supraglacial cryoconite sample had 27% Ascomycota, 22% Basidiomycota and 2.2% Chytridiomycota, and 49% of remaining library clones could not classified. Of the total 26 OTUs, 14 are related to Ascomycota cluster of sequences, including the genera Cladosporium, Aureobasidium, Tetracladium, Pseudeurotium, Fusarium, Penicillium, Simplicillium, Aspergillus. Cladosporium, which were absent in the subglacial sediment, were only present in supraglacial cryoconite sample. No clones related to Aureobasidium, a dominant group in the subglacial sediment (8.93%), were present in supraglacial cryoconite sample. The genera Aspergillus (17.86%) and Simplicillium (12.50%), which were rare in the supraglacial cryoconite library, were well represented in the subglacial sediment. In addition, Basidiomycota groups included relatives of Rhodotorula and Leucosporidium, of which the former well represented in the subglacial sediment (8.69%), the latter being the dominant group in the subglacial and supraglacial clone libraries. As a result, different glacial environments would harbor unique assemblages of microbes because they arose from different sources and/or undergo specific selective pressures in situ.
Key words: glaciers in the Tianshan Mountains; cryoconite; subglacial sediments; fungi; hylogenetics
DOI:10.7522/j.issn.1000-0240.2017.0088
收稿日期: 2017-01-06;
修订日期: 2017-06-05
基金项目: 国家自然科学基金项目(41271268; 40961002; 41140009); 冻土工程国家重点实验室开放基金项目(SKLFSE200903); 中国博士后科学基金项目(20100339)资助
E-mail: 508996158@qq.com
中图分类号: Q938
文献标志码: A
文章编号: 1000-0240(2017)04-0781-11
